当代生物哲学研讨会(二) 单细胞数据跨物种比较:理论,方法与未来
单细胞数据跨物种比较:
理论,方法与未来
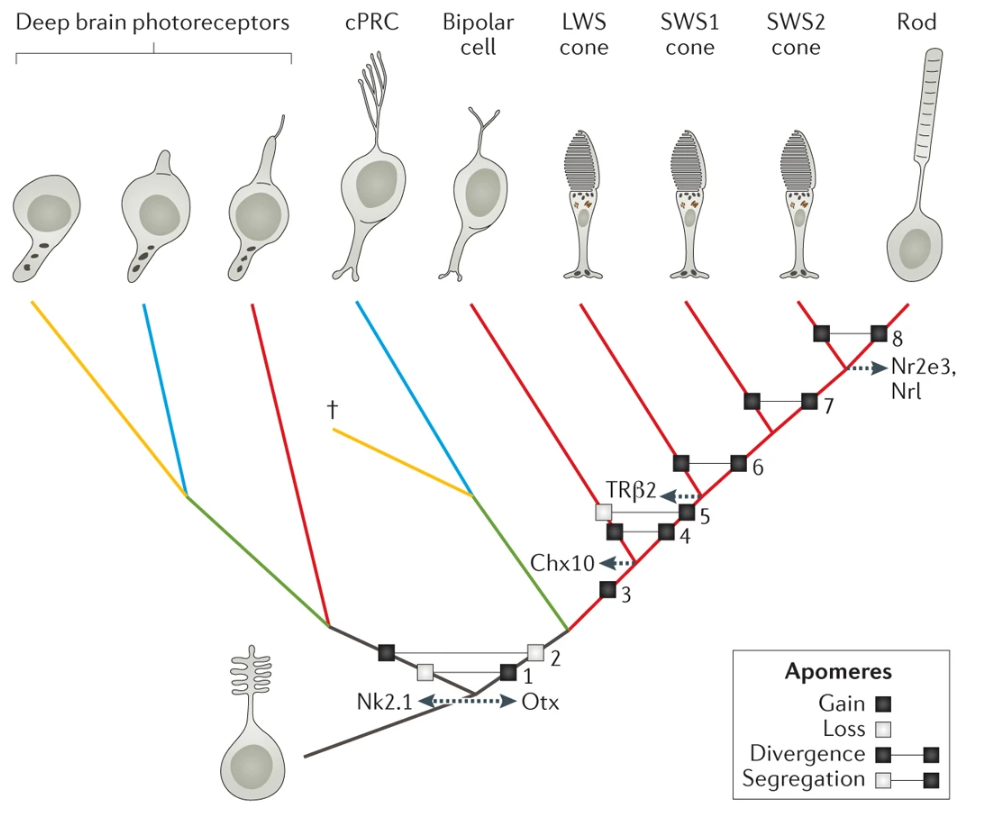
封面:两侧对称动物纤毛感光细胞的演化
(Arendt,2016,Nat. Rev. Genet. )
引子
为什么我们要关注细胞类型演化?
随着基因组数据的爆发,建立在分子演化理论基础上的比较基因组学方法在理清物种间关系方面提供了空前优势,但在解释复杂动物的形态演化问题上仍面临着巨大挑战。因为遗传学只能解释个体或群体之间的性状差异,而很难用来研究个体内部不同器官或者细胞基于基因表达调控的异质性。例如在细胞层面,人类的神经细胞与人类的心肌细胞共享几乎完全一致的基因组序列,但前者在功能和表型上却跟拥有完全不同基因组的果蝇神经细胞更加类似。我们可以发现,在复杂动物的演化历程中,不仅基因是独立的基本功能单元,不同细胞类型也同样能将各自独立的生物学功能稳定遗传给后代。因此,除了研究基因和基因组的序列变化之外,我们同样需要在细胞类型层面研究复杂性状的演化。
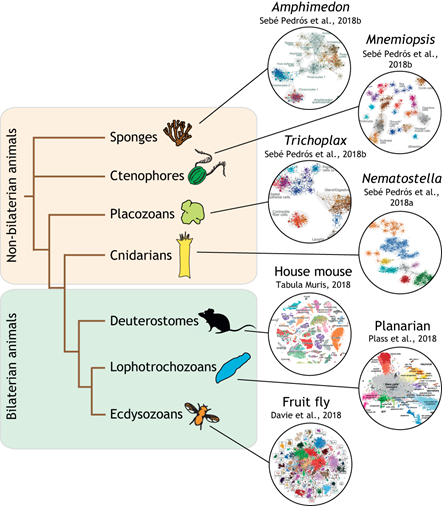
图1 蓬勃发展的跨物种细胞类型比较[1]
同源性是演化生物学的研究基础,只有同源的基因型或表型在跨物种比较时才有意义。但基于渐变论假设的新达尔文主义理论主要强调已有的同源表型如何演变,很难解释全新的组织或器官如何从无到有地产生[2]。事实上,人类、小鼠、果蝇、乃至水母都共享同一套发育调控基因集来完成胚胎发育[3],说明全新形态特征的出现并不意味着新基因的出现,而是意味着在旧基因的基础上搭建出了新的调控网络(gene regulatory network,GRN),这一系列新的GRN又指导了新的细胞类型与复杂性的形成。于是,我们能认识到,分子演化理论与细胞类型的演化理论在底层逻辑上拥有本质区别:不同基因之间是相对平权的,基因组中的一些基因不会成为另一些基因出现的前提条件;但细胞类型之间却存在着等级化的因果逻辑,即某些基础的细胞分化能力是另一些高级细胞分化能力的前提条件。例如脊椎发育要建立在脊索发育的基础上实现,所以脊椎动物要在最早的脊索动物的后代中出现;软骨发育是诱导硬骨发育的前提,所以最早的硬骨鱼在最早的软骨鱼的后代中出现。也就是说,与分子演化理论相比,细胞类型演化的视角更容易解释形态演化发生顺序背后的因果逻辑,这就是为什么我们有必要去建立一套完善的细胞类型演化理论。
(一)
细胞类型演化的理论框架
为讨论细胞类型的演化,首先需要思考如何定义细胞类型。从本质上来说,一种细胞类型是某类生物发育过程中可重现的具有固定谱系来源和发育潜能的细胞的集合[4]。这样的定义太过抽象,在实际中,人们通常会用可实现的技术手段逼近这一概念。例如,在仅有光镜和简单染色的时代,人们通过稳定的形态特征区分细胞类型,并用描述性词汇、研究者等为细胞命名,如用“浦肯野细胞(Purkinje cell)”指代小脑皮质中某一类神经元。随着分子生物学不断进步,人们对细胞的功能有了更深入的了解,基于功能的命名也随之涌现,如视杆、视锥细胞就指代视网膜中将光能转化为电信号的两类细胞。同时,分子标志物和流式细胞术的结合,促使学术界广泛使用细胞的“分子表型“进行交流,其中最经典的莫过于通过白细胞分化抗原(CD)建立的免疫细胞分类体系。近年来,各种单细胞测序技术越发普及,将细胞分子表型测量的广度与精度推向了新的高峰,各种基于聚类算法和先验注释的细胞类型描述手段应运而生。总体来说,人类对细胞状态的刻画由粗糙到精细,如今已经能实现分子层面的量化。根据我们目前对细胞运作原理的理解,具有相同转录状态的细胞足以被视为同一细胞类型。
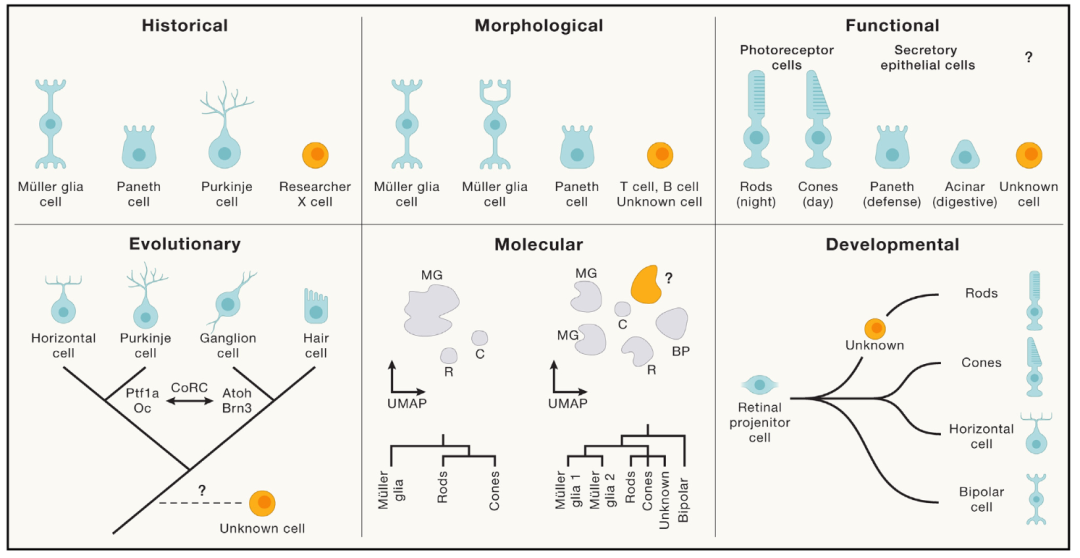
图2 主要的细胞类型定义方式。缺乏统一的框架妨碍了不同学科间的交流[4]。如今,单细胞技术的发展使得相关的定义与分析方法(molecular definition)成为囊括和拓展历史细胞类型的主流方式;但缺陷依然存在,如不代表真实发育轨迹、难以鉴定细胞类型同源性等。
然而,为了实现细胞类型的跨物种比较,仍有一个基本的理论问题亟待解决。那就是,怎样识别不同物种间同源的细胞类型?就像生物体任何层次的性状一样,真正的细胞同源性并非由功能或形态上的相似性所定义,而是由渐变论演化视角(descent with modification)下的连续性所定义[5]。由于每一代多细胞生物都要从受精卵开始,以一种相对固定的方式重建体细胞,因此有必要考察个体发育中细胞的命运决定过程,寻找可以用来推断连续性的保守成分。Eric Davidson指出,细胞分化本质上对应着一套相对独立的GRN激活与维持的过程[6]。然而,这一GRN中的成分并非平权的。一套核心的分化机制——通常由数个转录因子的调用网络所组成——比上游诱导分化的信号以及下游终末分化的表型更为保守。可以认为,这样的分化机制与细胞类型一一对应,它的改变,就会引起下游GRN和细胞表型的变化。因此,演化发育生物学家Günter Wagner称其为“核心调控复合物(core regulatory complex,CoRC)”,认为它是划分细胞类型的关键指标[7]。与基因的演化相类比,不同物种中共享CoRC的细胞可以被视为直系同源。同一个体内,在共同CoRC的祖先细胞类型基础上分化出有差异的细胞类型被称为姊妹细胞类型(可被视为旁系同源的细胞类型)。后者也是目前广为流传的新细胞类型起源的模型之一[7][8]。驱动细胞模块表达的大部分编码和非编码基因组信息仍然由两种姐妹细胞类型共享;只有一小部分对每种细胞来说都是特定的,这反映了个性化的萌芽,例如:组成脊椎动物视觉系统的细胞类型中,双极细胞被解释为纤毛感光细胞的姐妹细胞类型,它们是通过分工产生的,其中视杆细胞和视锥细胞“继承”了感光特性,双极细胞继承了轴突投射特性。
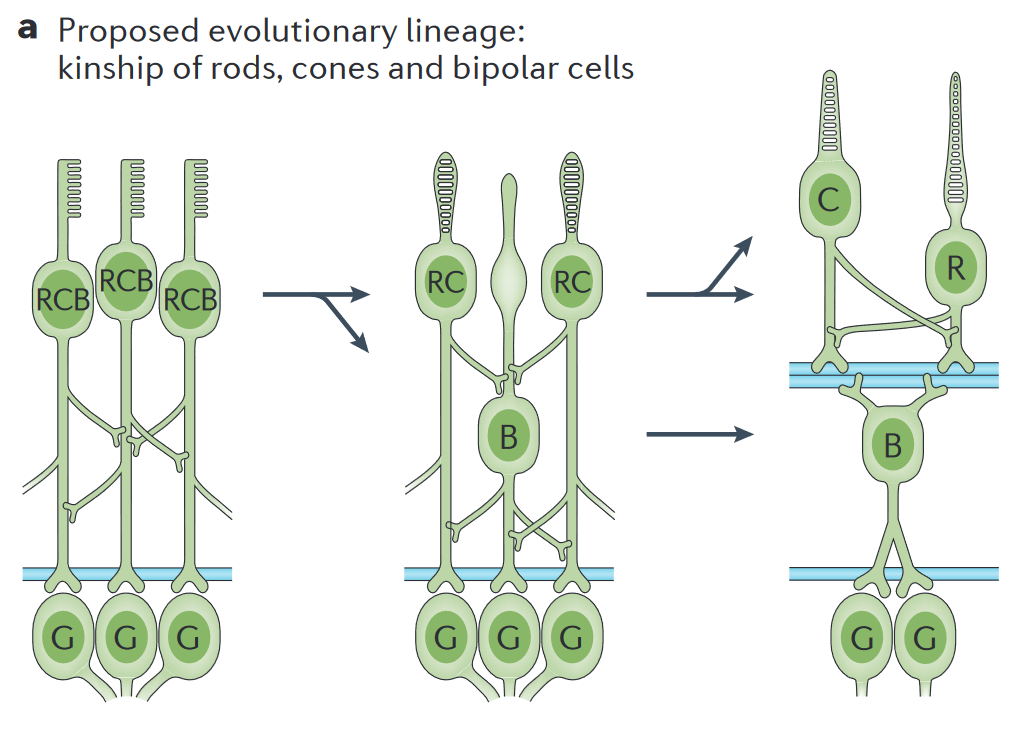
图3 脊椎动物视觉成像系统中细胞类型的演化机制。B,双极细胞(bipolar cell);C,视锥细胞(cone cell);R,视杆细胞(rod cell);RC, 视杆和视锥假想的演化前体;RCB,视杆、视锥、双极细胞假想的演化前体;G, 神经节细胞(ganglion cell)[7]。
根据这一思想,Wagner给出了细胞类型的演化定义:一种细胞类型就是一群因共享特异的CoRC而具有相对独立的演化能力的细胞[7]。然而,批评意见认为,这种划分方式需要对基因表达调控的网络有大量先验知识,因此缺乏客观性,也不利于对新的细胞类型的挖掘。真正无偏的,方便拓展的细胞类型定义,应当是数据驱动的[4]。在现有的实践中,最自然,最符合数据驱动标准的组织方式,包括基于细胞状态相似性的聚类算法,以及基于细胞分裂现象的谱系示踪技术。在接下来两节中,我们将首先介绍细胞聚类结果跨物种比较的实践与挑战,随后讨论在理论上我们如何整合细胞分化的知识与数据驱动的框架。
(二)
如何跨物种比较单细胞表达谱数据
遗传距离,选择压力,演化速率,基因型频率等一系列分子演化理论的核心概念都是在DNA或蛋白序列比对的基础上定义出来的。但是如何跨物种比较不同的转录状态则要比这复杂许多,这构成了当前研究细胞类型演化的巨大挑战。本节主要讨论跨物种整合单细胞转录组(scRNA-seq)数据的技术策略及其利弊。
任何跨物种细胞类型比较的第一步都是确定相关物种之间的全基因组基因同源关系。准确的基因同源性判断对于有监督的、以基因为中心的比较(例如,转录因子的使用)以及系统的跨物种分析(例如,细胞类型聚类或树构建)都是必不可少的[9]。重要的是,细胞类型比较必然涉及大的多基因家族,其系统发育关系和基于同源基因的分类难以解决,并且通常涉及谱系特异性扩展(例如,转录因子,离子通道或粘附蛋白)。因此,同源推断对于明确解释基因表达矩阵整合中的同源/同源关系是必要的,例如将一些分析限制为一对一的同源。
然而,大多数单细胞比较分析大多限制在近缘物种之间,在更长的演化距离和跨门的比较具有挑战性,主要有两个原因。首先,基因调控程序在演化过程中多样化,减少了细胞类型特异性基因表达模式的相似性。其次,复杂的基因演化史导致远缘生物共享很少的一对一基因同源物。因此,一些方法放宽了直系同源性(orthology)的限制,利用被定位细胞之间的表达相似性来推断旁系同源(paralogy)基因的相对贡献,从而有效降低基因特征的维度实现在遗传距离较远的物种间比较单细胞转录组数据[10]。
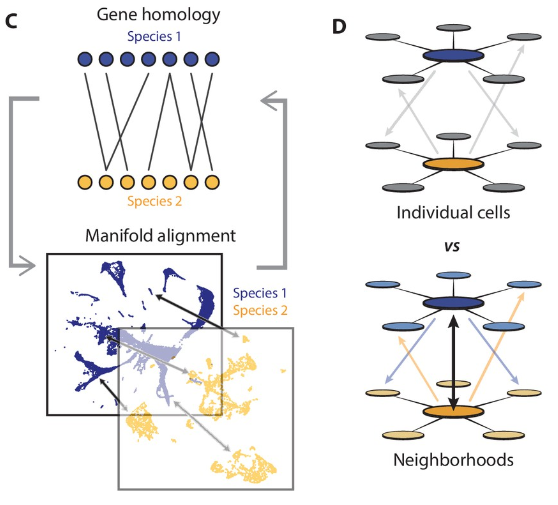
图4 一种基于更广泛同源基因(而非局限于1:1直系同源基因)表达相似性的跨物种scRNA-seq整合方案[10]
跨物种整合策略总体上分为单细胞水平直接整合,在细胞类型水平跨物种整合,以及在细胞状态(cell state)水平跨物种整合。比较直观的想法是直接在单细胞水平跨物种整合。然而这样的方案非常容易直接受到批次效应的影响,导致来自同一物种的不同细胞类型倾向于汇聚到一起。例如,在不同物种之间甚至是同一物种的不同年龄状态下,都会经常发现两个批次各自的中位数基因表达量有巨大差异,这为鉴定细胞类型的同源性带来了巨大的系统性偏差噪声。于是在单细胞水平整合的策略很难扩展到多个物种,尤其是在面对有很大系统发育距离的物种上时。
第二种策略是首先解析每个物种的细胞类型图,然后跨物种比较不同直系同源的细胞类型的总表达。通过聚类分析分出若干个在表达模式上互相独立的细胞群,并根据已有的生物学知识分别将这些群注释为一个个独立的细胞类型。这种策略允许在不同级别的聚类粒度上进行比较(例如,比较广泛的细胞类型或细胞类型亚群),并使其更容易解释非唯一或冗余的基因同源关系。混合策略包括单细胞整合,然后在整合空间中跨物种集群重叠。例如,“聚类优先”方法已经应用于神经细胞类型转录组的系统比较,揭示了小鼠和人类之间,甚至哺乳动物和爬行动物之间广泛的保守性[11]。
第三种策略则是建立在细胞状态水平(cell state)水平的跨物种比较上。细胞状态的概念与细胞类型来自于不同的分析策略。在面临有复杂细胞异质性的样本,例如某物种的全细胞图谱或发育过程中的胚胎时,由聚类分析划分出的群经常出现严重的内部异质性,即同一个群实际上可能是不同的细胞类型的混合体。因此,基于另一种目标的分析策略被开发出来,即追求每个组内无异质性优先而非组间的最大异质性优先,将所有单细胞划分为数百个组,每个组的细胞代表来自同一种细胞状态的多次重复抽样。这种分析策略允许不同细胞状态共享高度类似的表达模式,即代表着来自于同一种细胞类型的细微差异,可以用来分析几乎呈连续状态的表达模式。一般情况下,由数万细胞组成的数据可以倍划分为几十个细胞类型(群)或者数百种细胞状态(例如metacells),因此后者能更准确地将行使关键功能的稀有细胞类型鉴定出来[12]。由metacell等方法定义出的细胞状态被广泛应用在远缘物种间(跨门)的单细胞组学比较研究领域[13]。
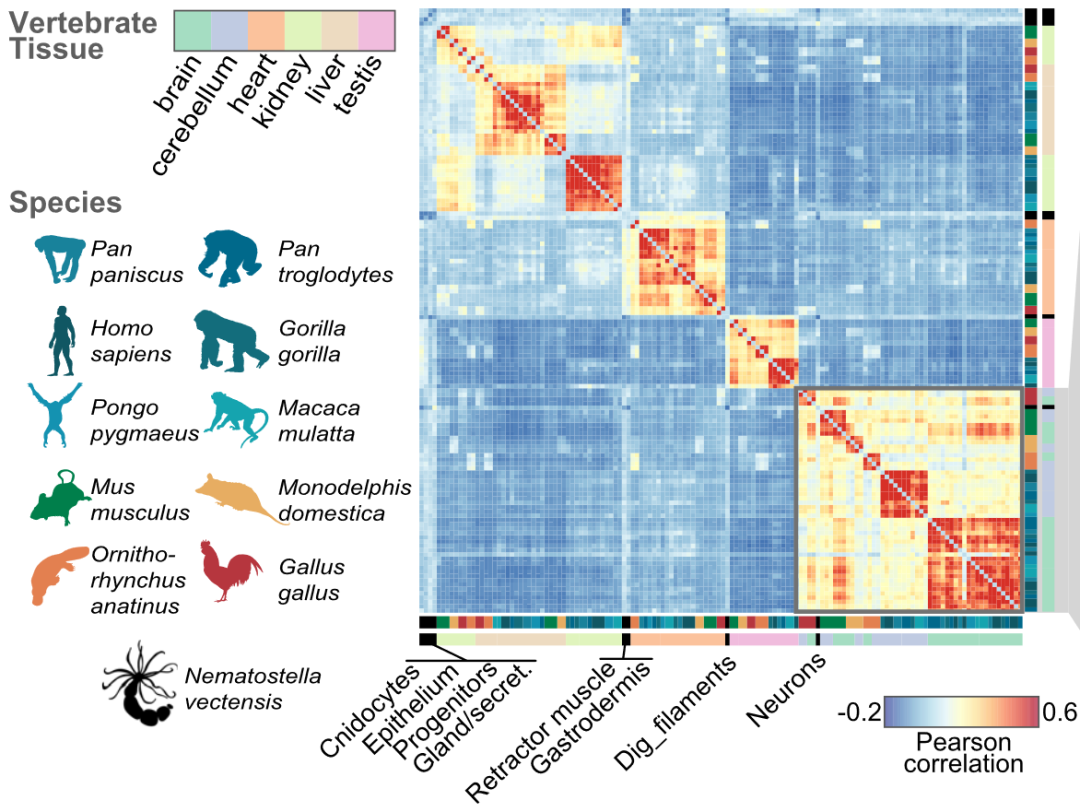
图5 Metacell水平比较跨门物种间的神经细胞类型同源性[14]
(三)
统一的框架:一个物种一棵“树”?
可以看到,现有的跨物种单细胞数据比较,多依赖于终末分化细胞类型间的相似性,与细胞类型演化的理论框架并不契合。这可能使我们失去诸如发现新细胞类型起源的机会。那么,未来的发展方向是什么?
或许,问题的实质是,如何将我们对细胞的知识组织在一个客观的,无歧义的,可拓展的框架之内。在这种框架下定义的细胞类型,将允许一切研究,包括通过跨物种比较寻找细胞类型的起源。
一个看似可行的方法是合并个体发育过程中多个时间点的组学数据,通过聚类和降维算法,形成详尽的单细胞发育图谱。然而,由于技术的局限(即一次观测后,“胞死不能复生”),这种图谱就像一张快照,仅能反映群体层面的多样性,而丢失了细胞个体的动力学信息。因此,它只能提供一个细胞状态变化方向的假设,难以回答某些经历快速变化的细胞类型的起源或不对称分裂的影响[15]。
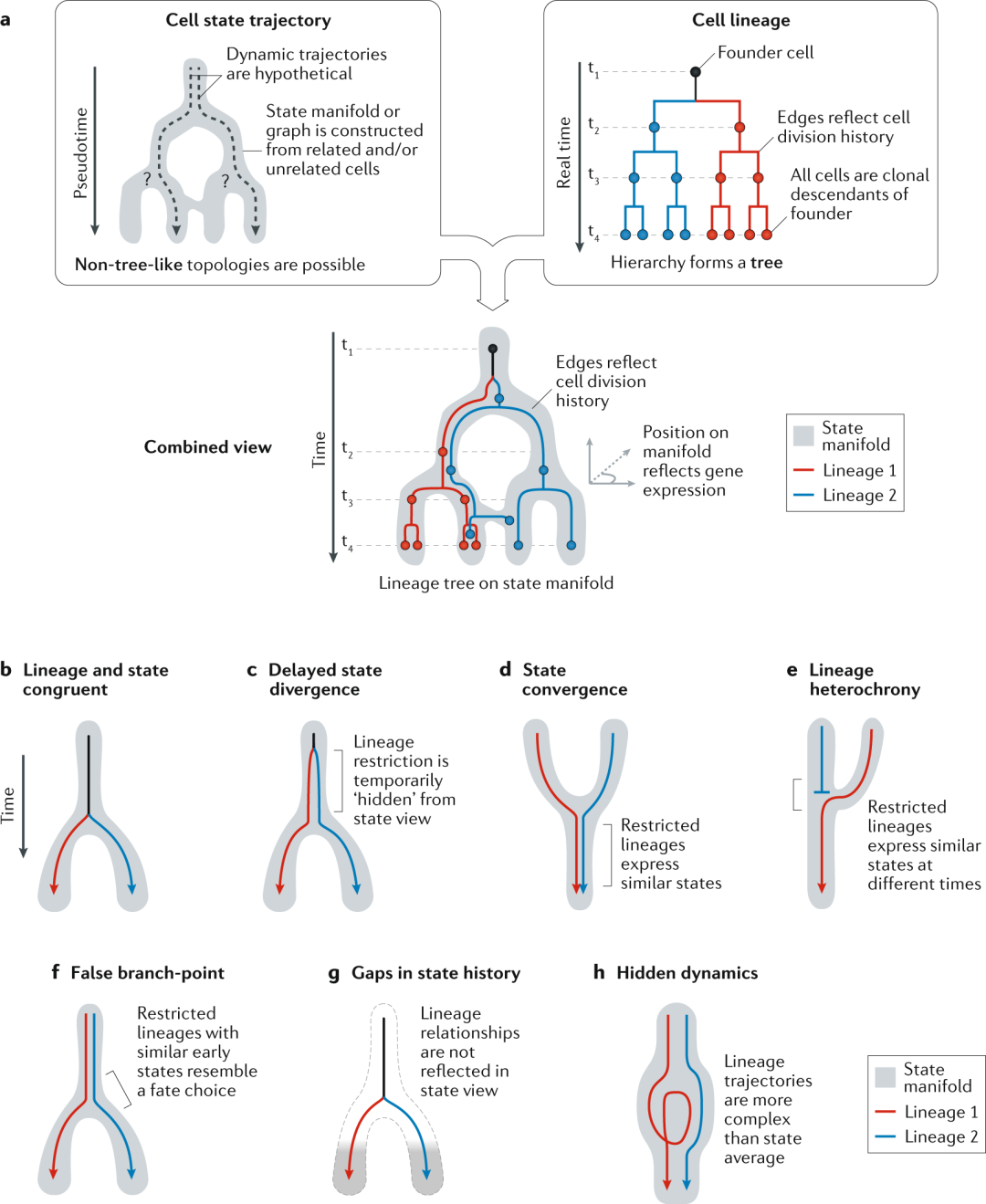
图6 状态相近并不意味着来源和动力学上的一致性。多细胞数据聚类降维得到的状态流形并不一定能反映真实的细胞分裂分化过程。但细胞谱系可以毋庸置疑地代表发育来源[15]
那么,如何为静态的图谱加上细胞分化的方向?一种解决方法是结合细胞谱系示踪技术,同时获取一个细胞的谱系信息和组学数据。谱系(lineage)指一个细胞分裂产生的所有子代。在多细胞生物的发育中,几乎所有细胞理论上都可以被置于以受精卵为根节点的二叉树上,因此谱系代表着无争议的发育起源信息。用于谱系示踪的实验手段有很多,除了早年的荧光标记,还可依靠转基因技术,在特定时期或条件诱导下在细胞的基因组上留下永久性的改变(“写入”),然后通过测序读取这种改变。此外,通过特定的实验设计与算法,还可以利用对DNA的持续写入甚至体细胞增殖中自带的突变,得到比“脉冲式标记”分辨率更高的细胞谱系[16][17]。
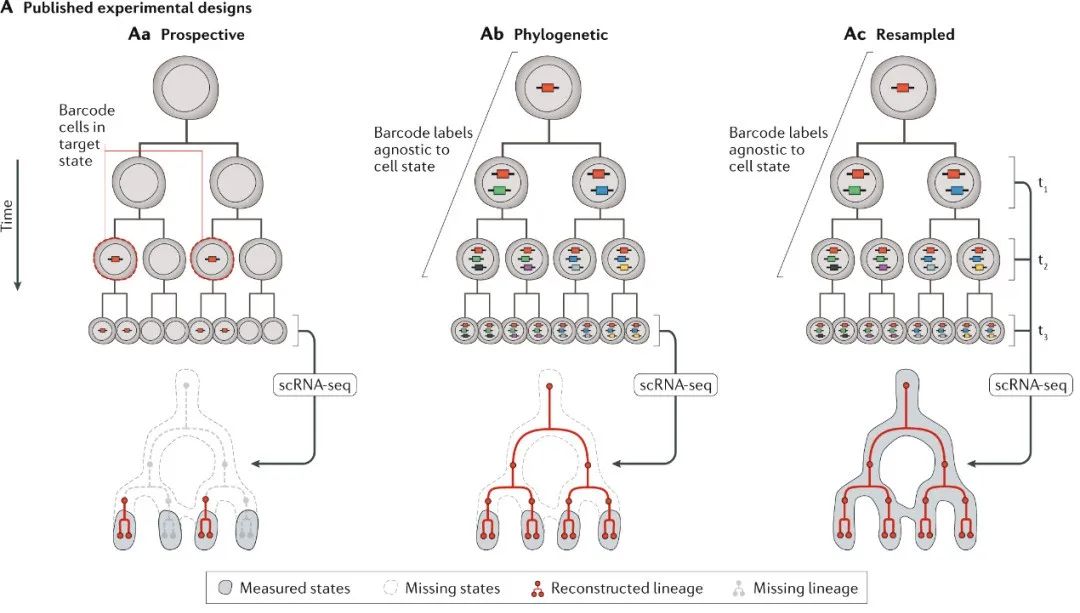
图7 左,静态谱系标记,在某一时间点标记全部或特异的细胞;中,动态谱系标记,持续标记所有细胞,可以重建较为完整的细胞谱系;右,动态谱系标记,且在多个时间点进行单细胞测序,可以重建具有完整谱系信息的状态流形[15]
未来,更先进的算法或许能直接从大规模动态谱系标记和组学数据中重建细胞的谱系关系,同时推测的祖先细胞的表达谱。那么理论上,我们可以确定一个个体丛生到死所有主要的细胞谱系,同时还有它们转录组模式的变化范围。事实上,在非常简单的物种,如秀丽隐杆线虫中,类似的树已经从幻想走入现实——因为这些动物有着极其精确且固定的细胞分裂模式和身体结构[18]。

图8 听起来确实是有点科幻的狂想?分析这么大的数据量,树形图设计者做得到吗(误)
基于上述思想,单细胞领域的先驱之一Jay Shendure认为,由于后生动物同一物种的个体发育过程具有高度可重复性,因此可以整合个体正常发育中所有细胞的谱系和组学数据,为每个物种建立一棵”一致性个体发育树(consensus ontogeny)”[4]。随后,我们可以使用客观的标准,如机器学习的最大信息增益,将细胞树的不同节段定义为不同细胞类型[19];也可以往这棵“树”上添加病理、衰老等不同状态下的单细胞数据。由此,纵览整棵“树”,我们可以说出每一种细胞类型的历史状态和当前状态,并预测其可能的未来走向。这种对细胞分化轨迹的完整识别将极大地推动针对CoRC的数据挖掘与评估,并深化我们对细胞类型的物种间同源性与演化规律的认识。
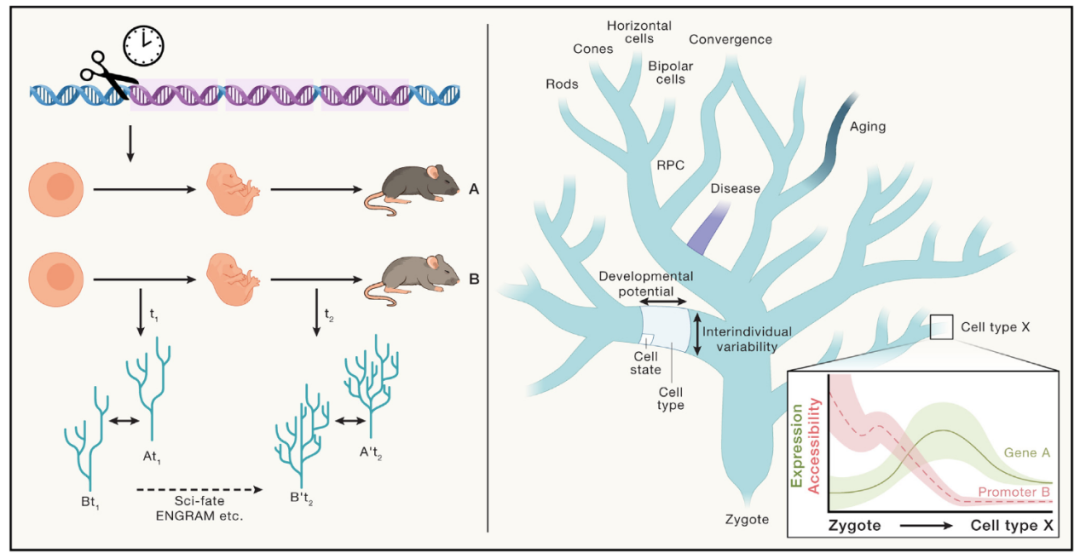
图9 左图,在构想中,构建“一致性个体发育树”的过程将包含:理想状态下从受精卵开始,标记每一次细胞分裂的谱系示踪;在不同发育时间点,对细胞谱系和转录组的同时测定;通过实验设计和算法组装不同时间点获得的单细胞数据;分析不同个体的数据,得到“一致发育树”。右图,“一致性个体发育树”的概念图,注意它并不是严格的树形结构,还存在不同谱系细胞表型的“趋同”[4]
总结
对于多细胞动物演化的研究来说,细胞类型的跨物种比较与基因序列比较同样重要。随着近年来单细胞组学技术的发展,这种比较在技术上变得可以实现。然而跨物种比较单细胞数据目前仍处于初级发展阶段,缺乏对一系列普适性概念的准确定义,也缺乏一个组织与分析数据的统一框架,未像分子演化理论那样形成一套完善理论。因此,理论的构建、概念的准确定义与统一,对当前的细胞类型演化研究而言迫在眉睫。结合细胞谱系示踪、单细胞组学数据构建“一致性个体发育树”,以及基于细胞分化调控关键基因的同源性识别原则,为细胞类型演化领域的发展提供了一种可能的思路。
参考文献
1. Sachkova, M. & Burkhardt, P. Exciting times to study the identity and evolution of cell types. Development 146, dev178996 (2019).
2. Pigliucci, M. & Müller, G. B. Evolution, the extended synthesis. (MIT Press, 2010).
3. Carroll, S. B. Evo-Devo and an Expanding Evolutionary Synthesis: A Genetic Theory of Morphological Evolution. Cell 134, 25–36 (2008).
4. Domcke, S. & Shendure, J. A reference cell tree will serve science better than a reference cell atlas. Cell 186, 1103–1114 (2023).
5. Wagner, G. P. The developmental genetics of homology. Nat Rev Genet 8, 473–479 (2007).
6. Davidson, E. H. & Erwin, D. H. Gene Regulatory Networks and the Evolution of Animal Body Plans. Science 311, 796–800 (2006).
7. Arendt, D. et al. The origin and evolution of cell types. Nat Rev Genet 17, 744–757 (2016).
8. Arendt, D. The evolution of cell types in animals: emerging principles from molecular studies. Nat Rev Genet 9, 868–882 (2008).
9. Quest for Orthologs consortium et al. Standardized benchmarking in the quest for orthologs. Nat Methods 13, 425–430 (2016).
10. Tarashansky, A. J. et al. Mapping single-cell atlases throughout Metazoa unravels cell type evolution. eLife 10, e66747 (2021).
11. Hodge, R. D. et al. Conserved cell types with divergent features in human versus mouse cortex. Nature 573, 61–68 (2019).
12. Baran, Y. et al. MetaCell: analysis of single-cell RNA-seq data using K-nn graph partitions. Genome Biol 20, 206 (2019).
13. Tanay, A. & Sebé-Pedrós, A. Evolutionary cell type mapping with single-cell genomics. Trends in Genetics 37, 919–932 (2021).
14. Sebé-Pedrós, A. et al. Cnidarian Cell Type Diversity and Regulation Revealed by Whole-Organism Single-Cell RNA-Seq. Cell 173, 1520-1534.e20 (2018).
15. Wagner, D. E. & Klein, A. M. Lineage tracing meets single-cell omics: opportunities and challenges. Nat Rev Genet 21, 410–427 (2020).
16. Kester, L. & Van Oudenaarden, A. Single-Cell Transcriptomics Meets Lineage Tracing. Cell Stem Cell 23, 166–179 (2018).
17. Farzadfard, F. & Lu, T. K. Emerging applications for DNA writers and molecular recorders. Science 361, 870–875 (2018).
18. Packer, J. S. et al. A lineage-resolved molecular atlas of C. elegans embryogenesis at single-cell resolution. Science 365, eaax1971 (2019).
19. Veleslavov, I. C. & Stumpf, M. P. H. Decision tree models and cell fate choice. http://biorxiv.org/lookup/doi/10.1101/2020.12.19.423629 (2020) doi:10.1101/2020.12.19.423629.










