Science专刊论文解读:亲缘更近但某些基因更远
我们都知道,人类是灵长类动物的一个分支,和类人猿、猴类等具有亲缘关系,但是远近有别。但有意思的是,亲缘关系更远的类群在某些基因上看起来却比较近的类群更相似。比如虽然黑猩猩是人类最近缘的现生物种,但人类基因组上有15%的区域却与大猩猩更相似。导致这一现象的原因是因为不完全谱系分流的演化遗传过程。近日,浙江大学生命演化研究中心张国捷团队与丹麦奥胡斯大学Mikkel H. Schierup团队共同合作,利用全基因组数据,对29个灵长类祖先节点的基因的不完全谱系分流现象进行了分析,回答了一系列相关问题。该研究是灵长类基因组计划的阶段性成果之一,已在著名学术期刊《科学》(Science)上以“Pervasive incomplete lineage sorting illuminates speciation and selection in primates ”(不完全谱系分流在灵长类演化过程中的影响以及其和选择作用的关系)为题发表。
产生“迷雾”的不完全谱系分流
作为人类的近亲,灵长类的演化过程与我们自身的起源息息相关。在灵长类的演化过程中,什么时候发生了什么事情,一直是演化研究关心的重点。物种在一代又一代的繁衍过程中,遗传物质DAN在向下传递的过程中会已一定的概率发生突变,带有有利突变的个体会因为更适应环境而产生存活下来,产生更多的后代,而这些有利突变也在群体中迅速扩散开并被固定下来。
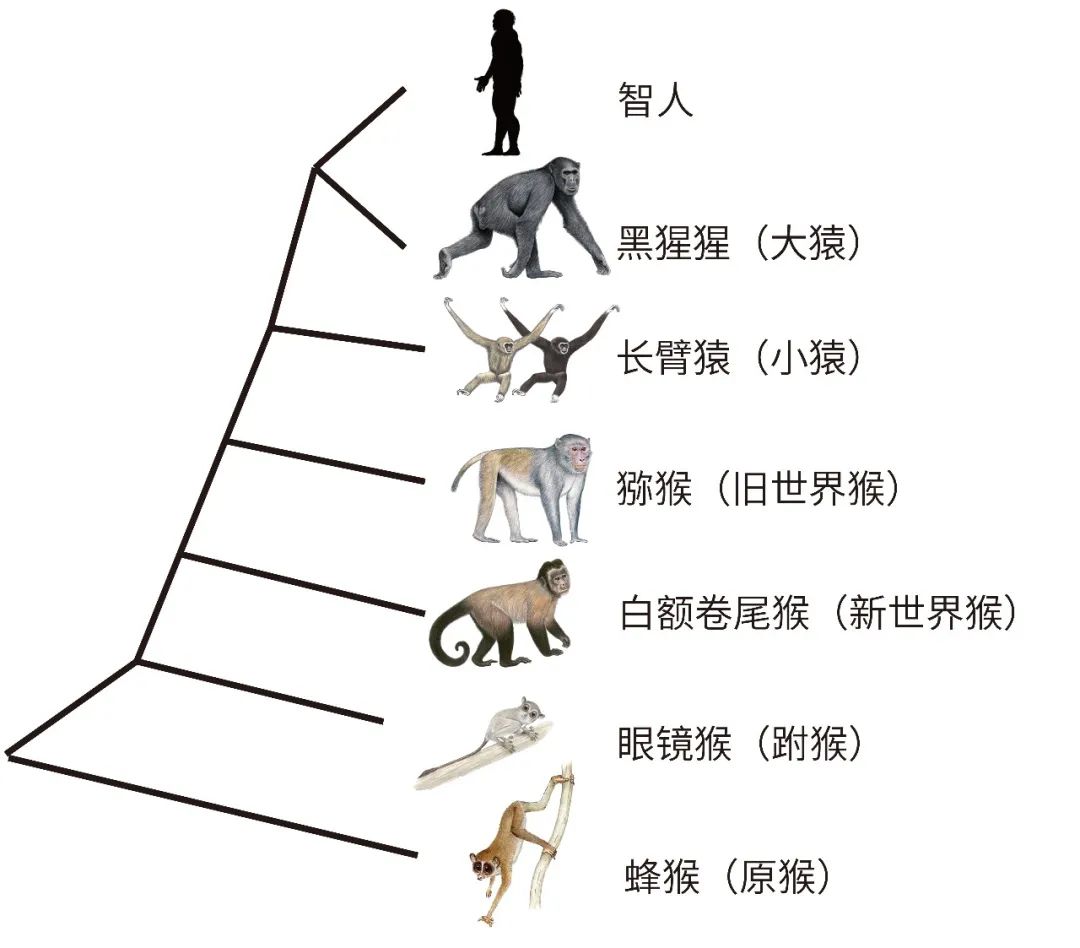
灵长类的系统发育关系简图,图片由李芳绘制(张国捷课题组 供图)
但是,还有一些突变会被随机继承,随着时间的流逝最终在群体中被固定下来或者丢失。但如果有些突变在被固定或者丢失前,也就是同一个基因同时存在多个基因型的时候,群体在短时间内发生多次物种分化,则这些多态性可能被后代随机继承,也就是会导致亲缘关系更远的物种因为继承了同一种多态性反而在序列上更相似,这种现象就属于不完全谱系分流(incomplete lineage sorting,ILS,或不完全谱系分选)。
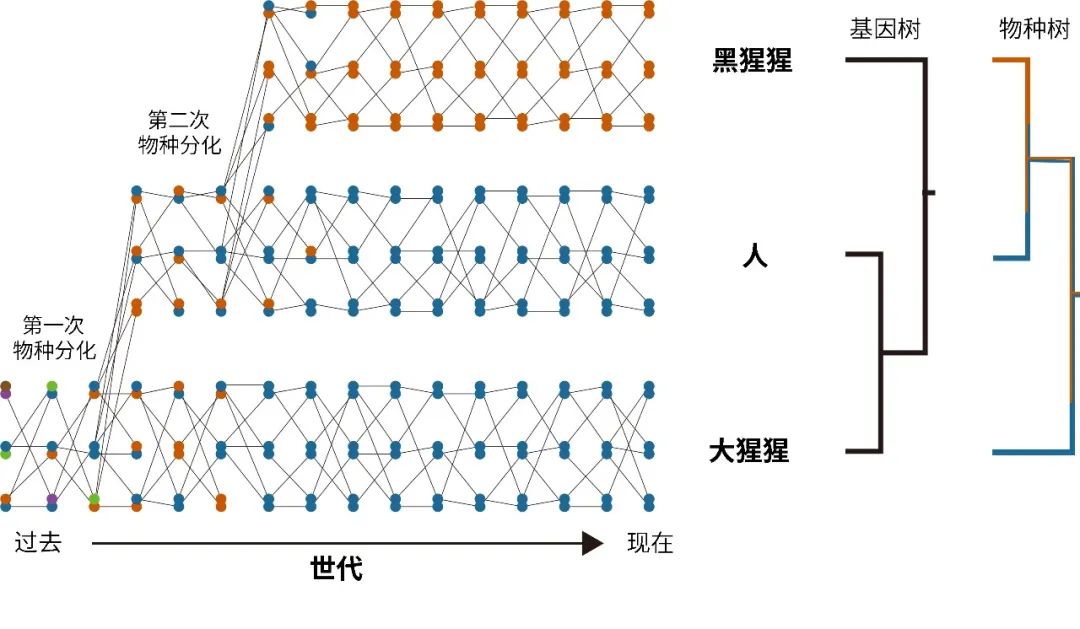
不完全谱系分流造成人、黑猩猩和大猩猩在基因树和物种树上不一致的示意图(冯少鸿绘)
不完全谱系分流作为一种演化上的现象,在多个物种中都被观察到过。以人、黑猩猩和大猩猩为例,我们知道,相较于大猩猩,人与黑猩猩的亲缘关系更近。如上图所示,每组两个圆点代表一个个体,每个圆点代表一个基因。三者共同祖先的群体在某个基因上具有较高的遗传多样性,也就是具有许多不同的基因型。而大猩猩在第一次物种分化后形成,随着时间的推移,大猩猩的群体最终固定了蓝色的基因型。人和黑猩猩的共同祖先群体则继承了橙色和蓝色两种基因型,然而在第二次物种分化的时候,人可能最终随机固定了和大猩猩一样的蓝色的基因型,黑猩猩最终随机固定了橙色的基因型。这样一来,我们就会观察到人的一些基因组序列跟大猩猩更相似,而跟黑猩猩差异更大,尽管黑猩猩才是与人最近缘的物种。事实上,在人的基因组里约超过15%的基因组区域,是跟大猩猩更相似的。
类似的现象在有袋目的演化过程中也被观察到过,基因组上有>50%的区域发生了不完全谱系分流,这使得只用部分基因组序列构建物种亲缘关系时会出现互相矛盾的结果。而整个灵长类的演化历程中,基因组上哪些区域,在什么时候曾发生了ILS,这些区域又与哪些基因有关,影响了哪些性状?为什么有些区域会有更高/低水平的ILS,是哪些因素影响了ILS的发生?这些目前并没有系统的研究。
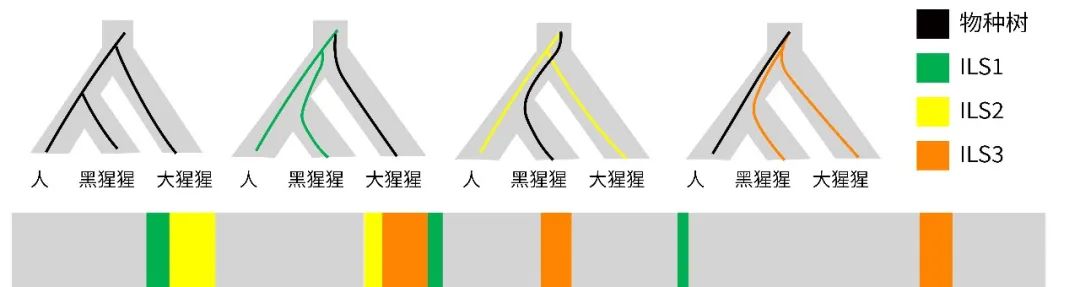
我们可以将基因组想象成一块块的马赛克,每一块都有自己独自的演化历程,而其中某些块则发生了不完全谱系分流。(李芳绘)
追溯灵长类演化过程中的不完全谱系分流事件
为了弥补这领域的空白,浙江大学张国捷团队,与丹麦奥胡斯大学Mikkel H. Schierup团队共同合作,基于全基因组数据,利用DNA演化的隐马模型(coalHMM),对29个灵长类祖先节点的ILS现象进行了分析。对于同一个基因在两个现存物种中不同序列,向上追溯其共同的祖先序列,该过程称为溯祖。而这一模型根据多物种溯组原理,可以推测基因组上每个位点最可能的演化历程,以及不完全谱系分流发生的概率。
研究发现,不同的时间点,灵长类基因组上有5%至64%的区域发生了不完全谱系分流,这说明在灵长类的演化历程中,不完全谱系分流在某些时间曾产生了较大的影响。在基因组上,不完全谱系分流片段的平均长度在100-1000个碱基对(bp),这意味单个基因,特别是那些长度超过20kbp的基因,很可能包含了多种不同的演化历程。因此,用部分基因组数据构建演化树时,会出现相互矛盾的结果,尤其是在长臂猿,猕猴和狒狒等中,高水平的不完全谱系分流正是一直以来这些物种演化关系难以确定的原因。
同时,coalHMM通过估算祖先群体大小发现,大部分祖先的群体都比现存物种群体要大。鉴于体型和群体大小的负相关关系,灵长类祖先物种可能体型都较小,又或者群体较小的祖先物种已灭亡,现存物种都是群体数量较大的祖先的后代。该方法对灵长类物种分化时间也进行了重新估算,结果与已知的化石时间基本一致。说明这个方法可以在没有化石证据的情况下,只用基因组数据和某些群体相关参数也可以得到较为准确的物种分化时间。
不完全谱系分流的发生与选择压力有关
而基因组上哪些区域容易发生不完全谱系分流,与哪些基因和表型相关,也是演化研究关注的重点。从演化的角度看,在没有选择压力的条件下,不完全谱系分流在基因组上应该是随机发生的。但是,将29个祖先节点的不完全谱系分流数据整合可以发现,基因组上存在某些区域,在整个灵长类的演化过程中一直保持着较高或者较低的不完全谱系分流水平,这反应了这些区域曾面临不同的选择压力。例如与转录调节相关的看家基因PIAS3,在几乎29个祖先节点都表现出了较低的不完全谱系分流水平,而与免疫相关的CD1A基因,在整个演化历程中不完全谱系分流水平都较高。另外,灵长类物种肤色对社交行为和性吸引等都十分重要,呈现出高度的多样化,而与角质化相关的基因也表现出了较高的不完全谱系分流水平。
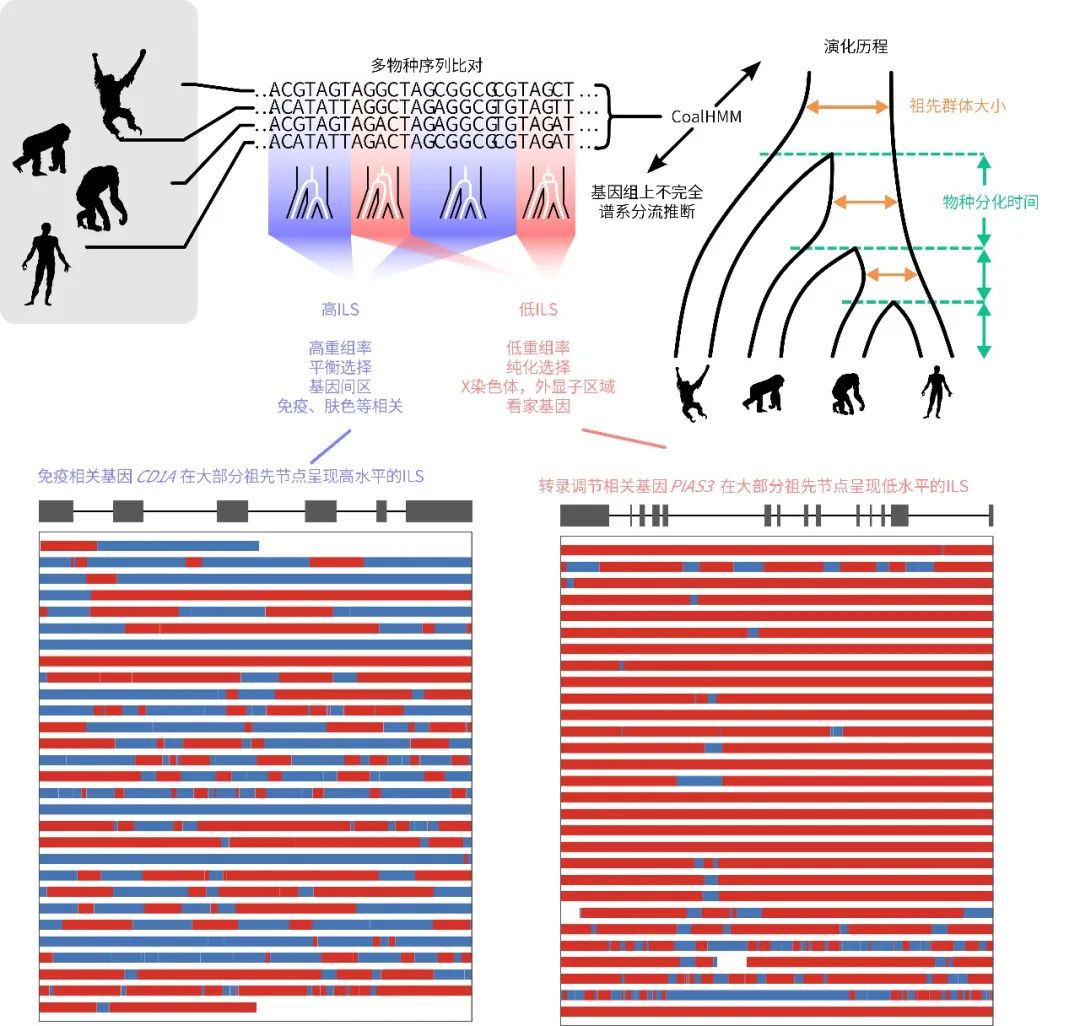
coalHMM在对基因组上发上不完全谱系分流的区域进行识别的同时,估算祖先群体大小和物种分化时间相关参数。高水平的不完全谱系分流区域通常与高重组率、平衡选择相关,低水平的不完全谱系分流则与低重组率,纯化选择相关。(Iker绘)
此外,除了选择作用外,基因或染色体的重组也可以对不完全谱系分流水平产生影响。选择作用会造成多态性的丢失,而重组会对多态性进行恢复。例如,人的2号染色体是由2条不同的染色体融合经过“端粒-端粒”融合而来,而融合的地方虽然在人类失去了端粒的高重组率特征,但在祖先中仍检测出较高的不完全谱系分流水平。










