SA论文解读:基因调控元件让灵长类彼此愈加不同
我们都知道,人属于灵长动物,我们的近亲还包括猴子、猩猩、长臂猿等灵长动物类群。由于较近的亲缘关系,灵长类彼此之间的基因组相似性很高,但彼此之间的表型差异却很大[1]。基因组上不同的区域有着不同的演化速率,有些区域非常保守,在几千万年的演化历史几乎没有变化,而有些区域则异常快速的变化,在某个类群或者物种里短时间内积累异常多的变异。这些超级保守或者快速演化的DNA序列,往往受到强烈的自然选择,提示这些序列具有重要的生物学功能。研究这些区域有助于回答灵长类不同类群之间的性状差异。近日,浙江大学生命演化研究中心张国捷教授团队和中科院昆明动物所吴东东教授团队联合在《科学进展》(Science Advances)上发表了题为“Lineage-specific accelerated sequences underlying primate evolution”(物种类群特异性快速演化序列对灵长类演化的意义)的研究论文。该研究是灵长类基因组计划的阶段性成果之一,通过大规模灵长类基因组的比较分析,鉴定了现存灵长类谱系所有关键演化节点的快速演化序列,并探讨了这些快速演化序列对灵长类动物多样性演化的功能意义。为深入了解灵长类动物,尤其是人类大脑特殊的发育模式提供了新的见解。
精确鉴定灵长类物种快速演化区域
早在1975年,M. C. King和A. C. Wilson就以非凡的洞见提出[1],人跟黑猩猩之间编码基因的差异非常少,基因调控元件的差异可以更好的解释人类和非人灵长类之间表型的明显差异。所谓的调控元件是基因附近的一段DNA序列,通过与转录因子和其它调控蛋白相互作用,增强或者减弱特定基因的时空特异表达模式,在生物形态发育过程中发挥着至关重要的作用。

形态各异的灵长类动物。图片来源:https://pixabay.com
与编码序列的变化相比,调控区域的改变通常不会导致基因表达的完全丧失,而是会对基因表达的水平或模式进行微调并可能影响到多个基因的表达,从而使得调控变异增加了生物的表型可塑性和适应性。在长时间的演化过程中,这些序列的高度保守性表明了其在功能上的重要性,而在这些超级保守的序列可能在某个特定物种类群中观察到的快速演化,说明这些区域可能在这个类群里曾经经历过强烈的正选择,使得该类群中发生了特有的有利突变,从而影响适应性新表型的产生。然而,对这些快速演化序列具体的影响范围和深度仍十分有限。2005年,黑猩猩基因组的发表为我们探究灵长类物种的演化以及现代人类起源提供了宝贵的机遇。仅时隔一年之后,由美国、比利时和法国组成的科学家研究小组,通过比较人类与黑猩猩基因组的差异,发现了在脊椎动物中演化非常缓慢,但在人类谱系中发生了显著变化的多个快速演化区域。其中差异最为显著的HAR1基因被认为是导致人类大脑皮层迅速增长的重要因素之一[2]。该文章同时也开启了人们对灵长类快速演化区域的探讨。2008年,Prabhakar等人发现一个在人类中演化极快的保守非编码DNA序列(HACNS1),相对于黑猩猩和恒河猴的同源序列来说,该元件作为基因表达的增强子有助于人类手指和肢体的发育[3]。2011年,Lindblad-Toh等人利用29只哺乳动物,发现了超过1000个人类快速演化序列,并揭示了与人类性状、健康和疾病之间的关系[4]。Girskis等人在2021年的一项研究中整合了前人超过3100个人类快速演化序列,发现近一半的序列是神经发育的增强子,并可能参与人类特异的神经和大脑发育[5]。
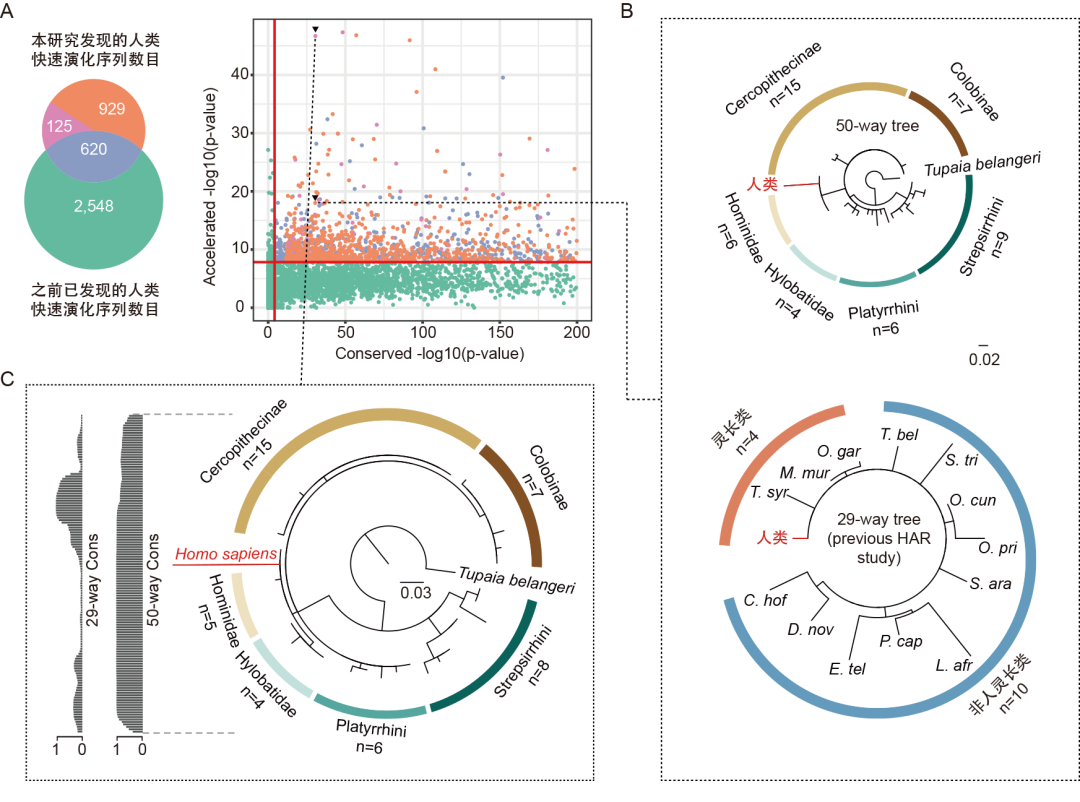
整合更多灵长类基因组提高了对人快速演化区域的鉴定能力。(A)本研究与前人在人类快速演化序列研究中的重叠和差异数目;(B)与前人研究相比,增加更多灵长类物种数目有助于对人类快速演化序列的检测;(C)与前人研究相比,我们找到了更多灵长类特异的保守区域 。( 毕旭鹏供图)
如今,本次研究通过利用49个灵长类高质量的基因组和成熟的比较基因组学手段,研究者对灵长类67个重要演化节点进行了大规模的快速演化区域研究,这也是迄今为止它们在灵长类动物中首次被精确识别和系统解析。各个谱系之间快速演化区域的数量差异非常大,范围从几百到几千不等。相比之下,超过70%的快速演化区域位于潜在的调控区域和长链非编码RNA中。这也表明这些区域在物种特有的表达调控和其它重要功能方面发挥了至关重要的作用。与此同时,在最新的研究中鉴定了超过1600个人类特有的快速演化区域,其中63%的区域为首次发现。与之前的研究相比,新找到的快速演化区域主要是由于灵长类基因组数量的差异所引起,从而影响了序列保守性和对人类加速演化信号检测的能力。通过增加更多近缘物种的数目并减少非灵长类动物基因组序列背景差异的干扰,可以发现更多灵长类特有的保守基因组区域,并使人类的加速信号相对于背景物种更为显著。
人类快速演化区域与大脑发育相关
和我们的近亲灵长类动物相比,人类的大脑占身体的比例在所有动物中名列前茅。先前已经表明,人类基因组中许多快速演化区域作为神经发育的促进剂,在神经元基因调控网络中发挥着重要作用[5]。
通过对人特有的快速演化区域相关基因的功能富集,我们同样发现这些区域与大脑和神经发育密切相关。例如,GBX2是一个参与菱脑原节正常发育的同源盒基因。在原肠胚形成和胚胎发育后期,与该基因相关的人特异快速演化区域可能促进了神经板的早期建立和大脑形态的正常形成[6]。此外,通过对前八个最为显著差异的人快速演化区域进行原位杂交实验,证实它们有助于人独特的大脑发育模式。
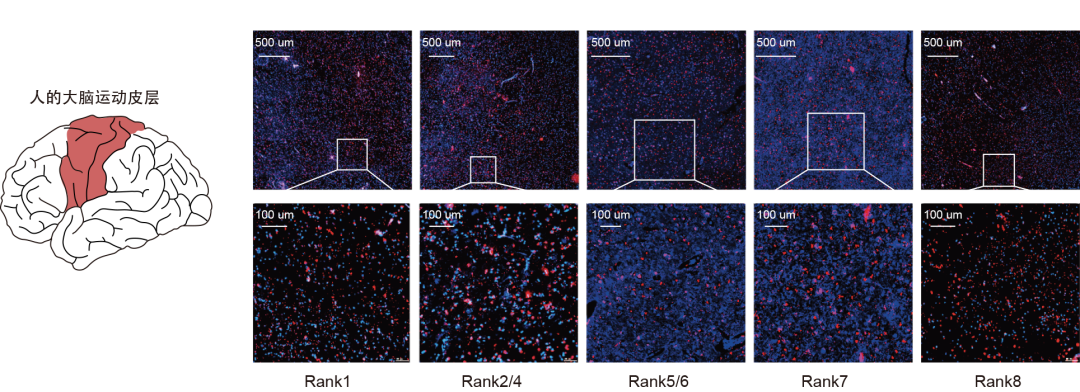
lncRNA在大脑皮层中的原位杂交表达结果。通过对本研究中首次发现的人类快速序列相关基因的原位表达分析,揭示了这些非编码基因在人大脑中发挥作用。(毕旭鹏供图)
森林“飞行猿”独特的前肢形态
长臂猿在所有灵长类动物群体中具有独一无二的运动方式,也就是通过用强壮的前肢来支撑自身体重,并依靠双臂的交叉摆荡在树冠层进行活动的方式。而这种运动方式形成了它们特殊的形态特征,如长长的前肢、发育良好的肩胛骨和强壮的肩关节,这些特征使得它们在快速摆动时可以进行大范围的运动。先前的研究表明一些与肢体发育的基因,如TBX5等,在长臂猿中经历了正选择,这可能与它们特有的前肢形态的形成相关[7]。
研究人员在长臂猿中发现了两个与肢体发育基因的调控区发生了快速演化,包括DLX5和EMX2。先前的转基因小鼠实验表明,EMX2基因参与了肩胛骨的形成[8],而DLX5敲除导致小鼠肢体结构存在多种发育缺陷[9]。为了检验这些区域在生物体内是否具有增强子活性,我们使用小鼠LacZ转基因报告实验进行了验证。结果表明这两个长臂猿特有的演化序列都能够在老鼠四肢以及肩关节区域发生激活,暗示了长臂猿该区域的突变可能造成了其前肢独特的发育。
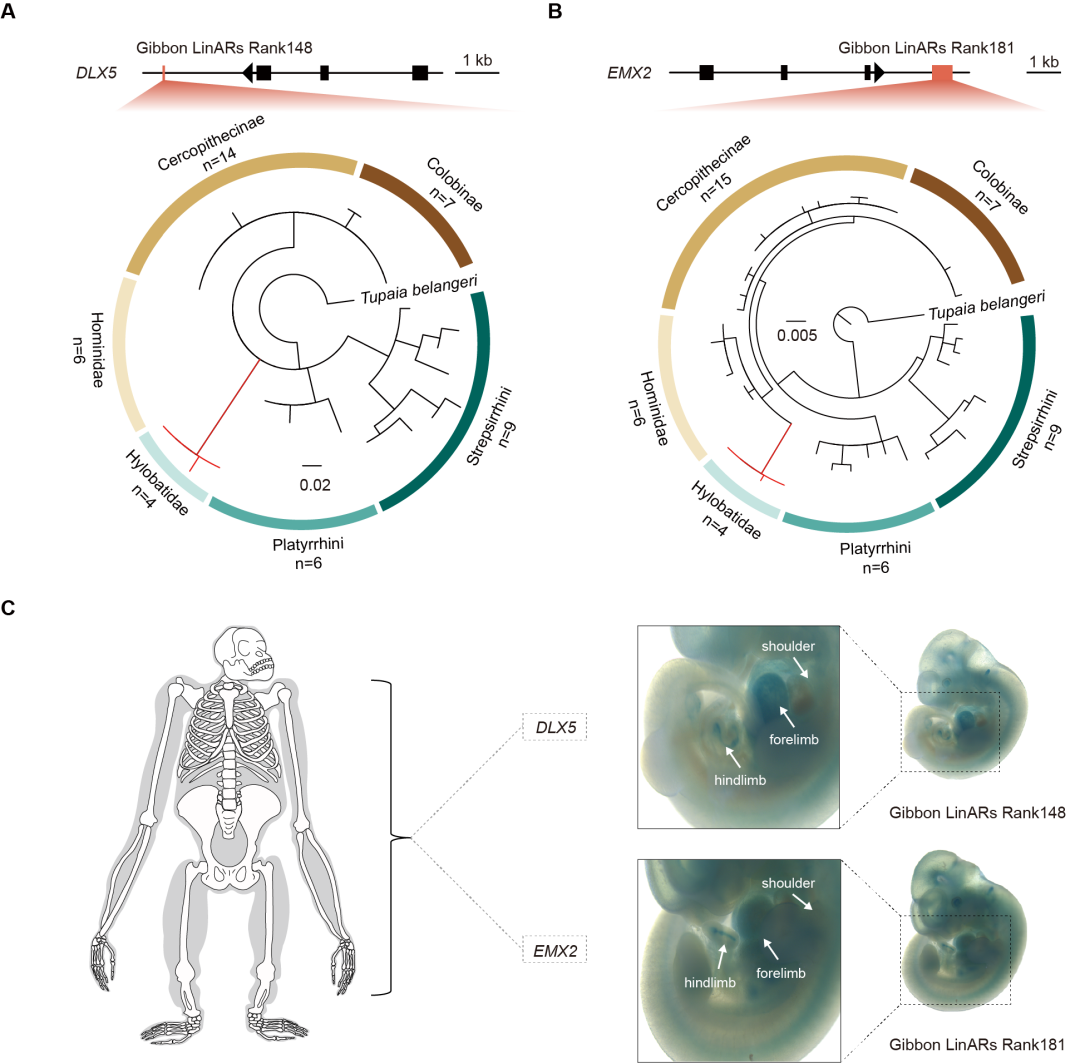
lacZ报告实验证实两个长臂猿特异的快速演化区域具有增强子活性。(A-B)两个长臂猿特异的快速演化序列分别位于DLX5和EMX2的基因下游区域。相对于其它灵长类,均显示出显著的核酸序列差异。(C)通过老鼠lacZ报告实验,揭示了这两个长臂猿特异的快速演化序列与四肢的发育相关。(毕旭鹏供图)
疣猴亚科独特消化系统的形成
疣猴别名为“吃叶猴”,主要食用树叶和种子。它们演化出高度特化的消化系统以适应其独特的饮食结构,这使得它们能更好地消化植物同时又能减少叶子毒素。此外,疣猴是唯一一种具有前肠发酵的灵长类动物,这让共生细菌在其高度特化的胃中分解植物成为可能。先前的研究表明,在疣猴粪便微生物群体中具有极高的细菌多样性,它们能够帮助对富含纤维物质的消化吸收[10]。虽然疣猴具有前肠发酵的消化系统,但其胃内细菌群落极其多样化的原因任尚不清楚。
研究团队在一个具有抗细菌、抗真菌和抗MRSA活性基因(SOD1)的内含子中发现了一段在其它灵长类中高度保守,并受到强烈的负选择,但在疣猴的共同祖先中发生多个碱基突变的区域。通过对人,猕猴和金丝猴不同组织的转录表达对比,发现SOD1基因在许多组织中广泛表达,但与人和猕猴相比,SOD1基因在金丝猴(疣猴的一种)中表现出较低的表达活性,特别是在胃,肝脏和肾脏中。进一步双荧光素酶报告实验证实在金丝猴中该序列的启动子活性显著低于人和猕猴,这表明SOD1在疣猴中的低表达可能是由这段快速演化区域所驱动。疣猴消化系统中较低的SOD1活性可能允许更多的微生物存活,从而帮助它们更好的消化叶子和种子等食物。
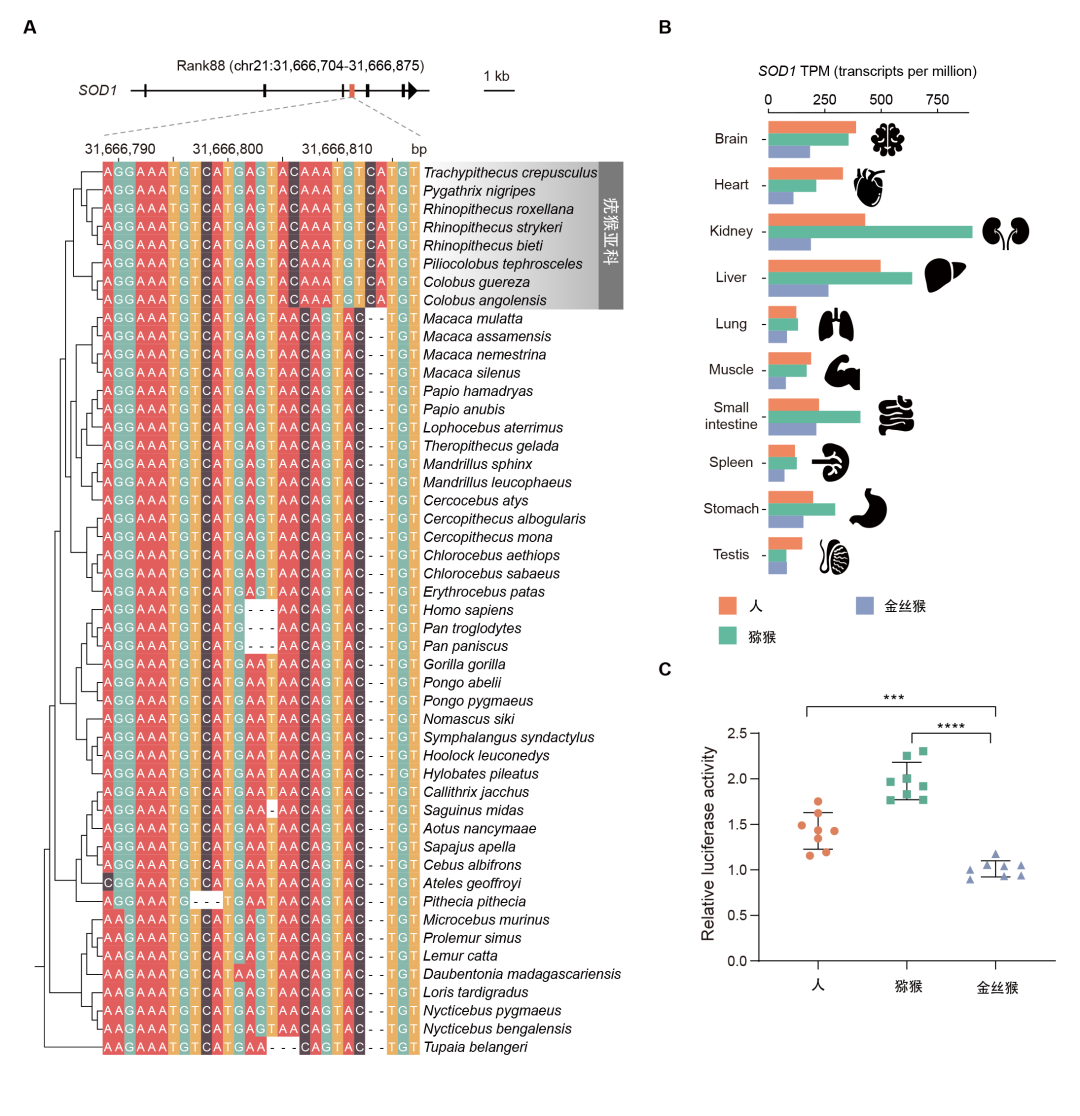
双荧光素酶报告实验揭示了疣猴该序列中较弱的启动子活性。(A)该序列在疣猴亚科祖先就已出现显著的核酸序列变化。(B)SOD1基因在金丝猴的肾脏,胃等多个器官的表达量较低。(C)实验证实该疣猴特异的快速演化序列具有较弱的启动子活性。(毕旭鹏供图))
综上所述,研究人员通过对多个灵长类基因组的比较分析,揭示了灵长类动物特有性状的形成与类群特异的快速演化序列之间的关系。通过对灵长类的研究,可以更好地理解人类的起源和演化过程的本质,并有助于我们了解人类生物学和行为学方面的基础知识,推进医学和神经科学等领域的研究与应用。
浙江大学生命演化研究中心张国捷教授、中科院昆明动物研究所吴东东研究员为该论文共同通讯作者。浙江大学生命演化中心毕旭鹏和周龙以及中科院昆明动物研究所张锦锦为共同第一作者。
参考文献
1. M. C. King, A. C. Wilson, Evolution at two levels in humans and chimpanzees. Science 188, 107-116 (1975).
2. Pollard K S, Salama S R, Lambert N, et al. An RNA gene expressed during cortical development evolved rapidly in humans[J]. Nature, 2006, 443(7108): 167-172.
3. Prabhakar S, Visel A, Akiyama J A, et al. Human-specific gain of function in a developmental enhancer[J]. Science, 2008, 321(5894): 1346-1350.
4. Lindblad-Toh K, Garber M, Zuk O, et al. A high-resolution map of human evolutionary constraint using 29 mammals[J]. Nature, 2011, 478(7370): 476-482.
5. K. M. Girskis et al., Rewiring of human neurodevelopmental gene regulatory programs by human accelerated regions. Neuron 109, 3239-3251 e3237 (2021).
6. S. T. Waters, M. Lewandoski, A threshold requirement for Gbx2 levels in hindbrain development. Development 133, 1991-2000 (2006).
7. Carbone L, Alan Harris R, Gnerre S, et al. Gibbon genome and the fast karyotype evolution of small apes[J]. Nature, 2014, 513(7517): 195-201.
8. F. Pröls et al., The role of Emx2 during scapula formation. Developmental Biology 275, 315-324 (2004).
9. R. F. Robledo, L. Rajan, X. Li, T. Lufkin, The Dlx5 and Dlx6 homeobox genes are essential for craniofacial, axial, and appendicular skeletal development. Genes & Development 16, 1089-1101 (2002).
10. B. Xu et al., Metagenomic analysis of the Rhinopithecus bieti fecal microbiome reveals a broad diversity of bacterial and glycoside hydrolase profiles related to lignocellulose degradation. BMC Genomics 16, 1-11 (2015).










