线粒体-细胞核的“对话”
真核动物细胞基本上都有两种基因组,线粒体和细胞核基因组。大约20亿年前,古菌和古菌病毒结合。在长期的演化过程中,自由生活的古菌和古菌病毒演化为特定的细胞器,古菌病毒(archaeal viruses)演化为细胞核,负责细胞的信息功能,比如能够调节途径和大部分转录和翻译;古菌(Alphaproteobacterial)演化为线粒体,特异地负责代谢功能[1]。从某种意义来说,基因组分工是内共生起源的结果。虽然线粒体和细胞核在基因组拷贝数、突变率、遗传模式以及复制和表达机制等遗传学的各个方面都有所不同[2],但为了维持正常的生命活动,仍然需要两个基因组之间的相互“交流”。
线粒体将遗传控制移到细胞核
在长期演化过程中,线粒体-核相互“交流”时,线粒体将遗传控制向细胞核转移。一方面,基因从线粒体转移到细胞核;其中许多基因已经进化出线粒体之外的功能。通过比较现存的真核生物的线粒体基因组的含量[3-5],研究人员已经推断出不同谱系中线粒体基因丢失事件的进化时间(图1)。这些分析将线粒体基因丢失的历史分为两个阶段。第一个阶段是在真核生物最后一个共同祖先(LECA)时期,在该阶段线粒体基因大量丢失;第二是一个异质的阶段,在时间和系统发育谱系中都有变化[2]。对于单个谱系来说,目前的线粒体基因含量是由长时间的停滞期和加速的基因丢失所决定的。例如,在两侧对称动物之前,谱系特异性的丢失和转移导致线粒体基因组只有13个蛋白编码的基因。另一方面,将核编码的蛋白质基因募集到线粒体中来协助组成氧化磷酸化复合体等,其中许多不是线粒体的基因[6]。线粒体蛋白质组的分析表明被募集到线粒体中的核编码的蛋白质基因主要是来源于以前存在的核基因,这些核基因将会行使新的功能。
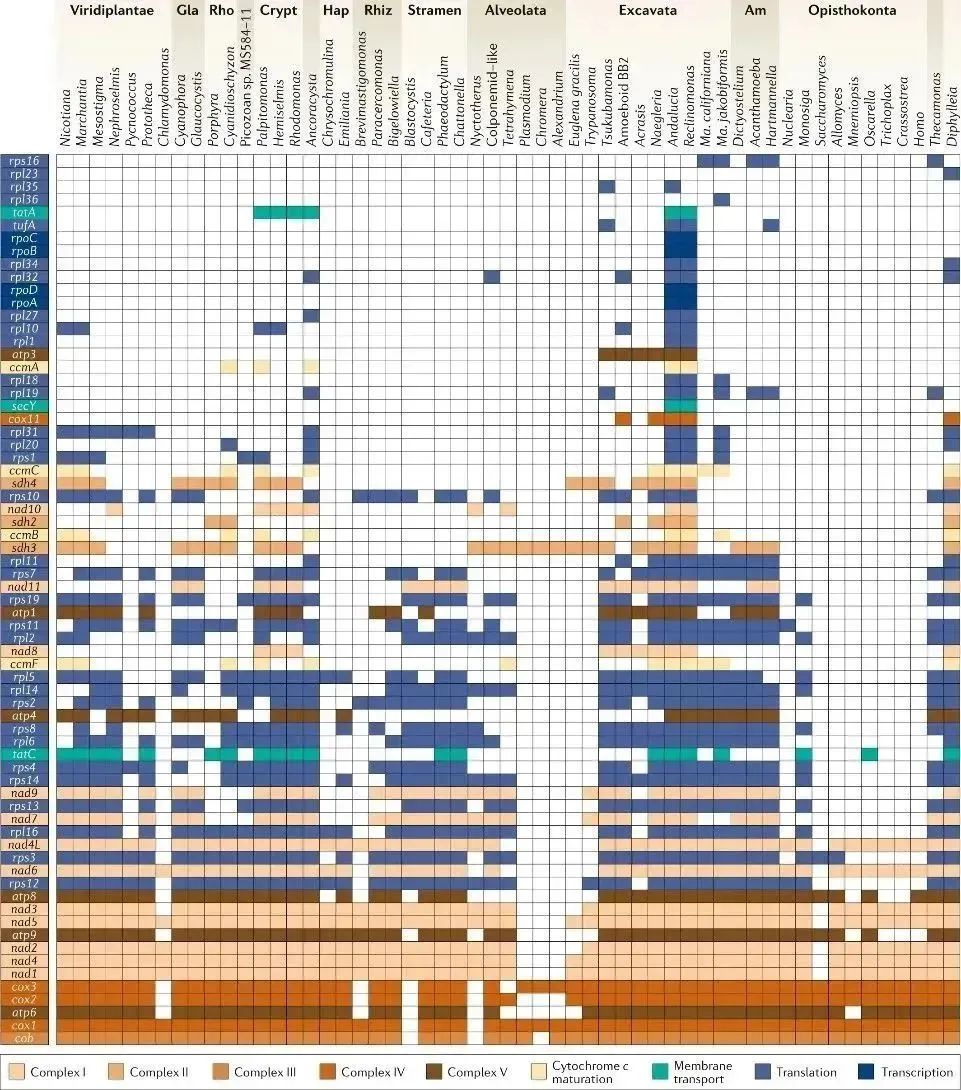
图1. 真核生物谱系中线粒体基因含量的变化
线粒体-核基因组共进化
线粒体和核基因组编码的蛋白质基因组成的氧化磷酸化复合体受到不同突变率,有效种群大小和遗传模式的影响,是线粒体-核共适应的标志。那么氧化磷酸化复合体中的线粒体编码的蛋白质基因(mtOXPHOS)和核编码的蛋白质基因(nuOXPHOS)之间是以何种共进化方式维持着氧化磷酸化途径呢?关于氧化磷酸化复合体共进化的假说有:核补偿共进化、协同共进化和中性变化驱动的共进化。
说起核补偿共进化[7],首先要先介绍一下缪勒氏齿轮(Muller's ratchet),它是指在没有重组的情况下(尤其是在无性繁殖的种群中),不可逆的有害突变积累的过程。核补偿共进化是核基因组进化以补偿线粒体基因组中的有害等位基因,从而保证线粒体不会失去功能,为缪勒氏齿轮悖论提供一个潜在的解决方案。核补偿共进化被认为有两种不同的形式:1)新的核编码氨基酸序列的进化,它能逆转或缓解线粒体基因组有害变化引起的特异性功能障碍;2)募集新的核编码蛋白质作为电子传递链复合物或复聚体的辅助亚基,有助于稳定并减轻线粒体基因组中有害等位基因引起的酶功能变化。虎斑猛水蚤(Tigriopus californicus)种群研究中与线粒体编码的蛋白质基因具有直接功能相互作用的核编码蛋白质(Complex Ⅰ, Ⅲ, Ⅳ, Ⅴ)显示出比不直接与线粒体基因相关的核编码蛋白质基因(Complex Ⅱ)拥有更高的进化速率(图2),为支持核补偿共进化假说提供了证据[8]。当然在其他物种中也有否定这个假说的例子[9]。因此,核补偿进化在某些真核生物谱系可能是一个很重要的驱动力,但不能认为它是真核生物中线粒体-核共进化的全部形式。
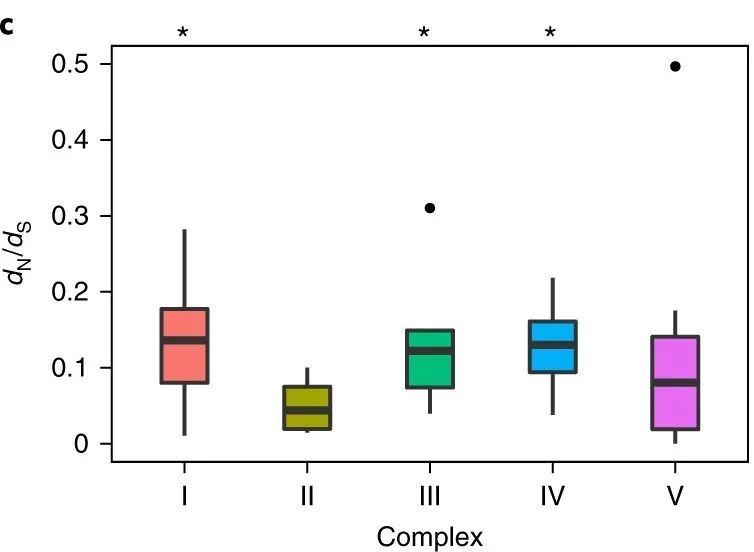
图2.比较氧化磷酸化中核编码的蛋白质基因的dN/dS
线粒体-核协同共进化是其中一个基因组的最初有益的变化会引起另一个基因组发生新的有益变化。线粒体-核协同共进化,维持几乎所有真核生物适应性的关键功能。由于自然选择或漂移,不同地区的种群可能具有不同的线粒体基因组序列。通过对北美洲西部的橙尾鸲莺属 (Setophaga) 中两种不同气候分布的杂交莺基因组分析,表明在两个不同区域杂交种中染色体5号上有一段高度分化的区域是与线粒体功能直接相关的基因所在区域,与线粒体基因组共进化[10](图3)。该研究表明与气候相关的线粒体-核选择可能是推动种群分化的重要力量。
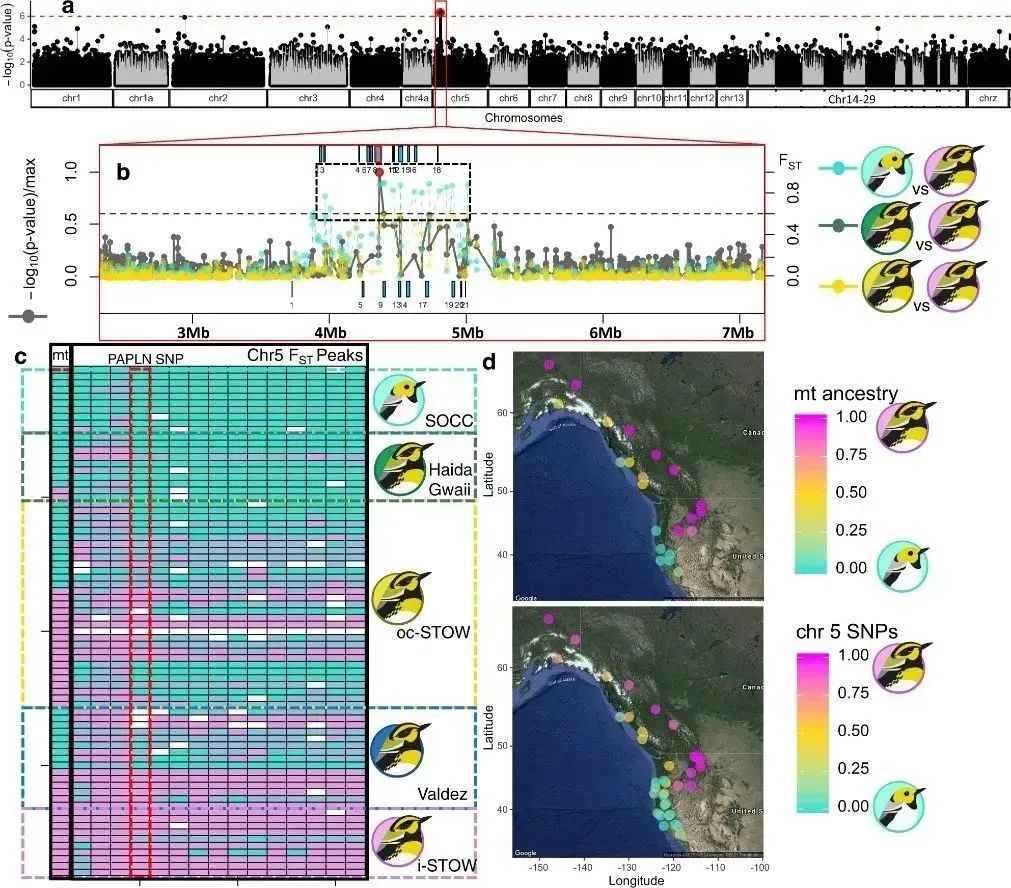
图3.橙尾鸲莺属种群中线粒体-核的高度分化区域一致
线粒体-核基因组之间相互“交流”的研究,不仅在真核生物的起源方面,而且在有性繁殖、性选择,适应以及物种形成等方面都有着非常重要的作用。氧化磷酸化复合体是研究分子共进化非常完美的模型,值得在种群、物种、细胞、分子等水平研究。
参考文献
[1] Cotton, J. A. & McInerney, J. O. Eukaryotic genes of archaebacterial origin are more important than the more numerous eubacterial genes, irrespective of function. 107, 17252-17255, doi:doi:10.1073/pnas.1000265107 (2010).
[2] Sloan, D. B. et al. Cytonuclear integration and co-evolution. Nature Reviews Genetics 19, 635-648, doi:10.1038/s41576-018-0035-9 (2018).
[3] Williams, T. A., Foster, P. G., Cox, C. J. & Embley, T. M. An archaeal origin of eukaryotes supports only two primary domains of life. Nature 504, 231-236, doi:10.1038/nature12779 (2013).
[4] Kannan, S., Rogozin, I. B. & Koonin, E. V. MitoCOGs: clusters of orthologous genes from mitochondria and implications for the evolution of eukaryotes. BMC Evolutionary Biology 14, 237, doi:10.1186/s12862-014-0237-5 (2014).
[5] Janouškovec, J. et al. A New Lineage of Eukaryotes Illuminates Early Mitochondrial Genome Reduction. Current Biology 27, 3717-3724.e3715, doi:10.1016/j.cub.2017.10.051 (2017).
[6] Wiedemann, N. & Pfanner, N. Mitochondrial Machineries for Protein Import and Assembly. 86, 685-714, doi:10.1146/annurev-biochem-060815-014352 (2017).
[7] Hill, G. E. Mitonuclear Compensatory Coevolution. Trends in Genetics 36, 403-414, doi:10.1016/j.tig.2020.03.002 (2020).
[8] Barreto, F. S. et al. Genomic signatures of mitonuclear coevolution across populations of Tigriopus californicus. Nature Ecology & Evolution 2, 1250-1257, doi:10.1038/s41559-018-0588-1 (2018).
[9] Weaver, R. J., Rabinowitz, S., Thueson, K. & Havird, J. C. Genomic Signatures of Mitonuclear Coevolution in Mammals. Molecular Biology and Evolution 39, doi:10.1093/molbev/msac233 (2022).
[10] Wang, S. et al. Signatures of mitonuclear coevolution in a warbler species complex. Nature Communications 12, 4279, doi:10.1038/s41467-021-24586-8 (2021).










