着丝粒——基因组的暗物质
# 01
着丝粒的发现
细胞分裂贯穿着生物体的诞生、成长、衰老和死亡,是一种受到精密调控且有序复杂的生物学过程。在真核生物中,染色体通过复制、凝聚、排列,与纺锤体微管相连,同一染色体的姐妹单体被物理地拉向相反两极,实现遗传物质的平均分配。
1882年,Walther Flemming在开花植物百合(Lilium)和两栖动物蝾螈(Salamandra)中描述了染色体在有丝分裂中的分离过程,并注意到每条染色体都有一个纺锤丝附着点,呈现出狭窄收紧的特点,他将其称为主缢痕(primary constriction)。后来,“着丝粒(centromere)”这一同义词逐渐用于指代主缢痕处的DNA(或指染色体)。而最初与之等同的“动粒(kinetochore)“则逐渐用来指代着丝粒DNA上组装的一系列蛋白质复合物结构,这一结构附着到微管上,协调染色体运动,并确保染色体的均等分离。
着丝粒的显著特征之一是其核小体含有H3组蛋白变体CENH3(哺乳动物中称为CENP-A)。CENH3能与H2A、H2B和H4形成特殊核小体,招募下游其他着丝粒蛋白,指导动粒组装,被认为是功能着丝粒的表观遗传因子。与正常组蛋白H3相比,CENH3演化较迅速,具有高度分化的N端和相对保守的组蛋白折叠域,在不同物种或亚种中出现了一定程度的适应性演化[1]。
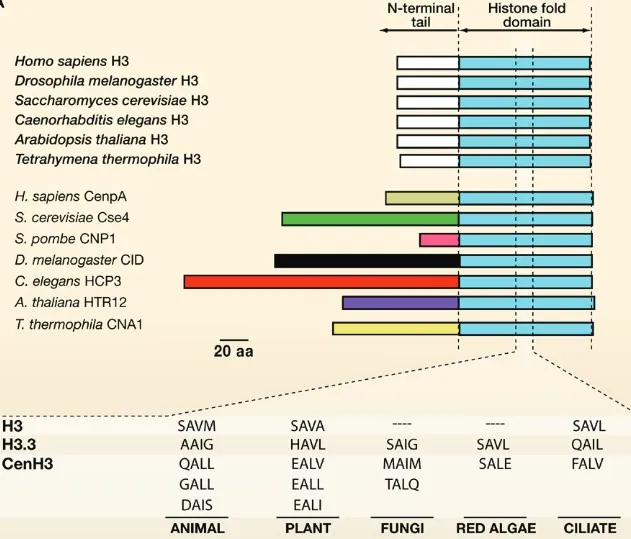
图2、不同物种中正常组蛋白H3与CENH3蛋白序列特征[1]
# 02
着丝粒的高度多样性
自最初的鉴定以来,着丝粒一直被认为是基因组中最神秘的部分。尽管着丝粒的功能在真核生物中高度保守,但在不同物种着丝粒序列的组织结构具有很大差异。
根据着丝粒分布,染色体大致可以分为单着丝粒型(monocentromere)、多着丝粒型(meta-polycentromere)和全着丝粒型(holocentromere)(图2)。单着丝粒是指着丝粒位于染色体的一个特定区域,在有丝分裂中能够观察到主缢痕的类型。绝大多数的真核生物,如人类、小鼠、果蝇、大多数植物和脊椎动物都属于这一类型。全着丝粒型是指在细胞分裂时着丝粒(纺锤丝牵引)的位置遍布整条染色体,缺乏主缢痕的类型。根据已报道的近800个全着丝粒物种的系统发育分析发现,由单着丝粒向全着丝粒的转变至少独立演化了13次,其中至少有四次发生于被子植物,多次在节肢动物和线虫中发生。而多着丝粒表现为延长的主缢痕和多个独立的着丝粒区域,近期在豌豆中观察到,类似于单着丝粒和全着丝粒类型之间的中间演化形式[2]。

图3、不同类型的着丝粒示意图 [3]
在单着丝粒类型中,不同物种着丝粒在序列组成和长度上同样高度可变(图3)。在酿酒酵母中发现的最小着丝粒约120bp,包含三个着丝粒决定元件(centromere-determining elements,CDE),能与CENH3特异性结合,是执行着丝粒功能的充要条件,被称为点着丝粒。而动物和植物的着丝粒通常由高度串联的重复序列和TE组成,长度可达到Kb至Mb,为CENH3和其他着丝粒蛋白提供了区域性的潜在结合位点,被称为区域着丝粒[4]。
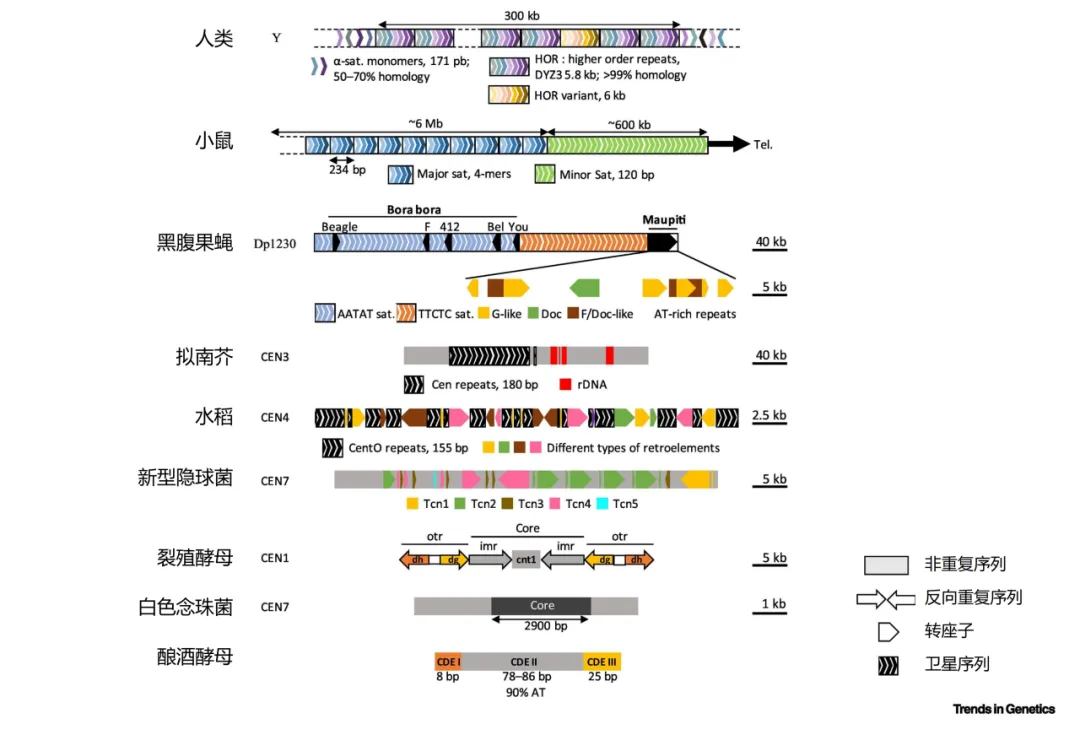
图4、真核生物着丝粒序列多样性[5]
构成着丝粒高度串联重复序列的最小基本单元称为重复序列单体(monomer)。不同物种具有不同的单体,例如人类的着丝粒主要是由171bp的α卫星序列重复串联组成,而家鼠(Mus musculus)的着丝粒和周边着丝粒序列则是由120bp的MiSat和235bp的Masat分别组成。有研究表明,在大约5000万年的时间跨度之后,不同物种单体序列几乎不再相似,整体缺乏保守性[6]。
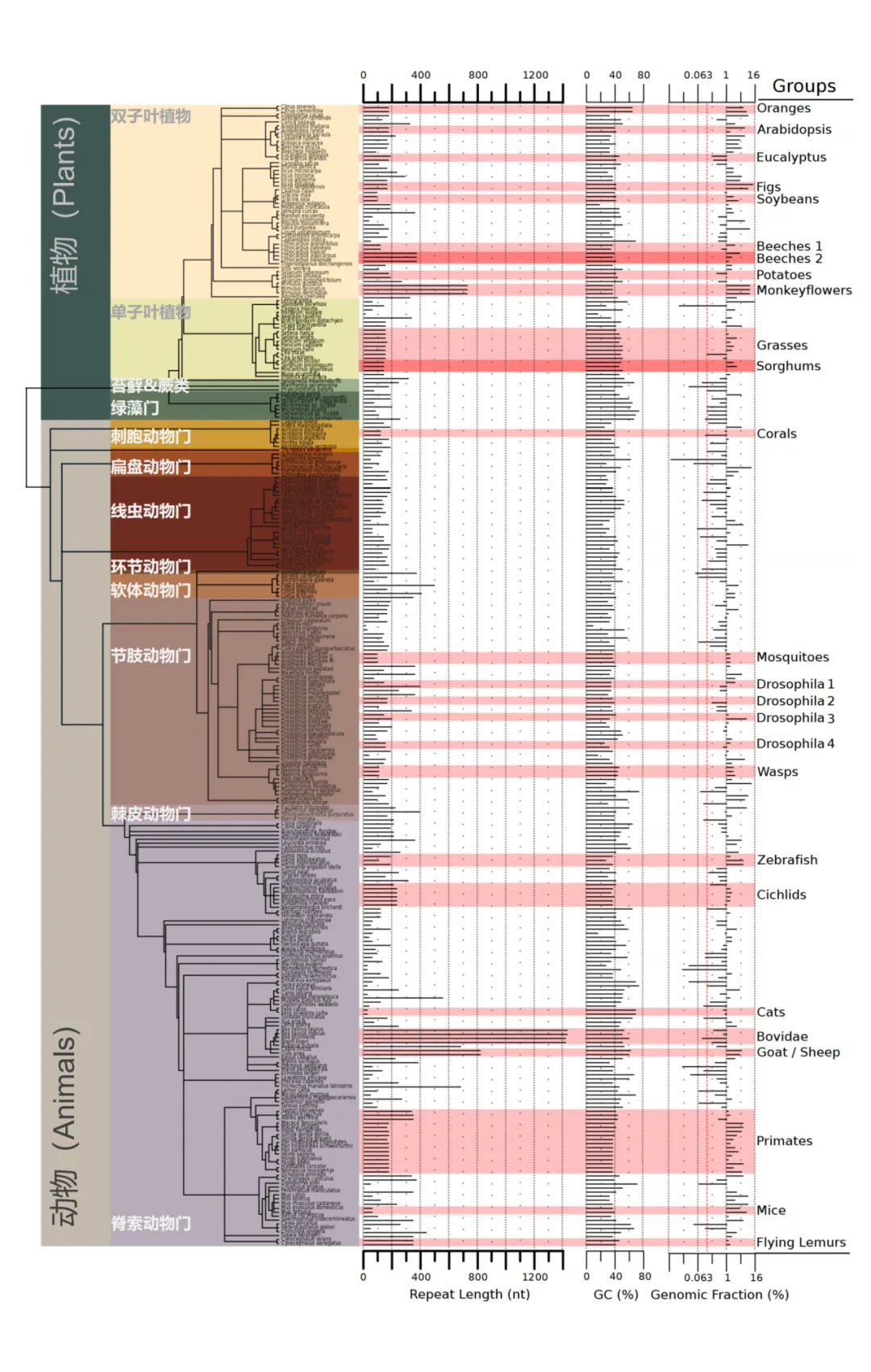
图5、282个物种(204属于动物,78个属于植物)之间的系统发育关系及(候选)着丝粒重复单体长度、GC含量和基因组占比(log2)。大约三分之一的物种,聚成26组(浅红色背景),这些组内的单体具有一定序列相似性,而组之外或组之间缺乏保守性[6]。
相比基因组的其他部分,着丝粒及相关蛋白为何演化得如此之快?Carmen Sapienza在2001年指出着丝粒可能在减数分裂驱动中扮演某种角色,并提出了着丝粒驱动假说。根据着丝粒驱动假说,在雌性减数分裂的过程中,由于只有一个单倍体细胞最后可以形成卵细胞,染色体的分离似乎并不遵循孟德尔定律,即不偏不倚地随机分离。一些“自私”的着丝粒,通过劫持染色体分离机制,提高了它们传递到下一代的几率。具体而言,着丝粒的DNA序列越长,其所结合的CENH3及其他着丝粒蛋白也会越多,这种“强“着丝粒则具有更大的几率进入卵子[7]。而同时,近期有研究表明,存在另一种机制,通过异染色质招募另一种着丝粒蛋白CENP-B会抑制染色体传递的偏差,使天平回归平衡[8]。研究者认为在漫长的演化历程中,自私因子与抑制因子相互制衡,开启了类似于免疫防御系统与病原体之间的“军备竞赛”。而这种快速演化可能是“军备竞赛”留下的重要证据。
图6、着丝粒驱动与抑制的两种平行途径[8]
# 03
着丝粒——疾病与衰老
演化过程中,着丝粒驱动的行为可能会带来适应性代价,例如染色体的错误分离引起的非整倍体配子造成的生殖细胞疾病。唐氏综合征是人类最常见的非整倍体疾病,由于存在额外的第三条21条染色体导致患者智力不足和发育延迟。除了已知的母亲年龄会增加患病风险外,着丝粒组织结构差异也可能在这一过程中发挥潜在作用。
一项唐氏综合征家族着丝粒研究发现患者携带的三个不同的21号染色体着丝粒单倍型在长度上相差11倍,最长和最短的单倍型都来自母亲,且最大的着丝粒呈现了模糊的CENP-A结合特征。由此看来,染色体着丝粒在大小和表观遗传差异上的不对称性可能会增加非整倍体的风险[9]。
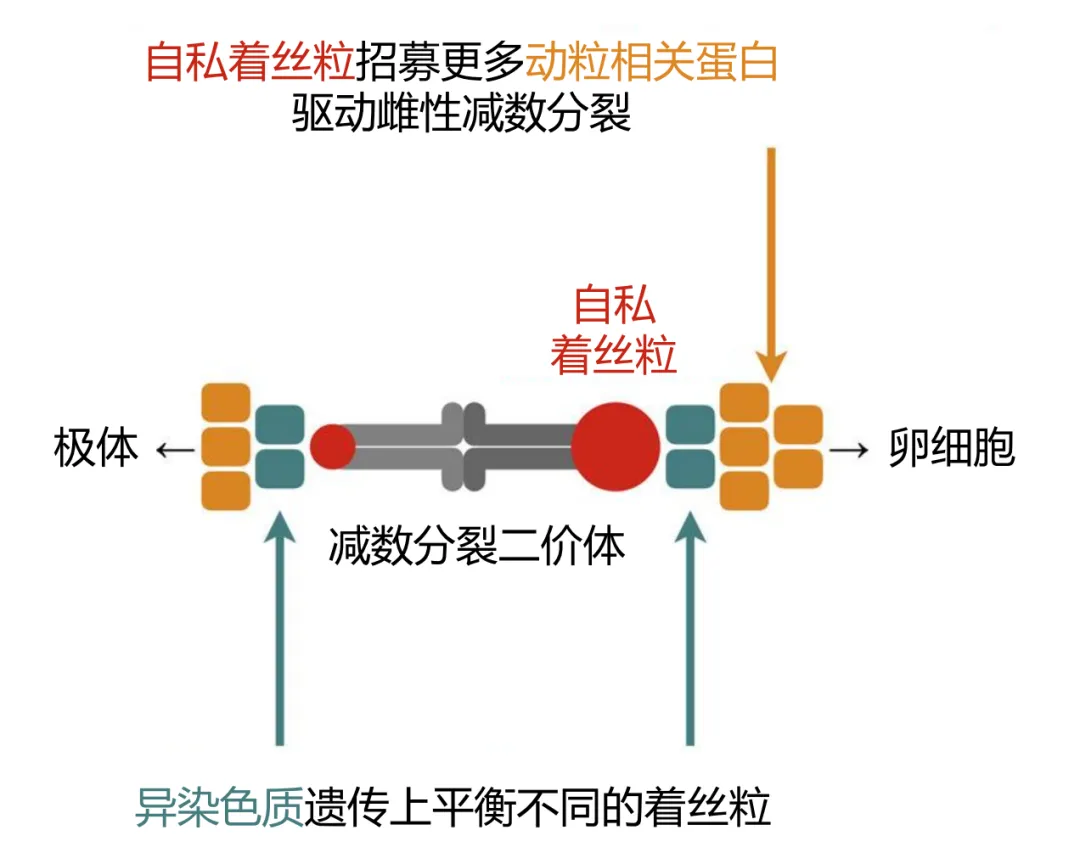
图7、唐氏综合征患者21号染色体着丝粒的遗传组成和表观遗传谱[9]
着丝粒断裂引起的染色体重排、非整倍体也常常在癌细胞与衰老细胞中观察到。利用针对着丝粒区域特异设计的qPCR方法,研究发现不同的癌细胞系与正常细胞相比表现出了广泛的异质性,包括α卫星阵列和HERV-K拷贝数量的显著减少[10]。在结直肠癌和腺癌的细胞系中,涉及(周围)着丝粒区域的染色体重排和断裂频率高达40-60%[11]。
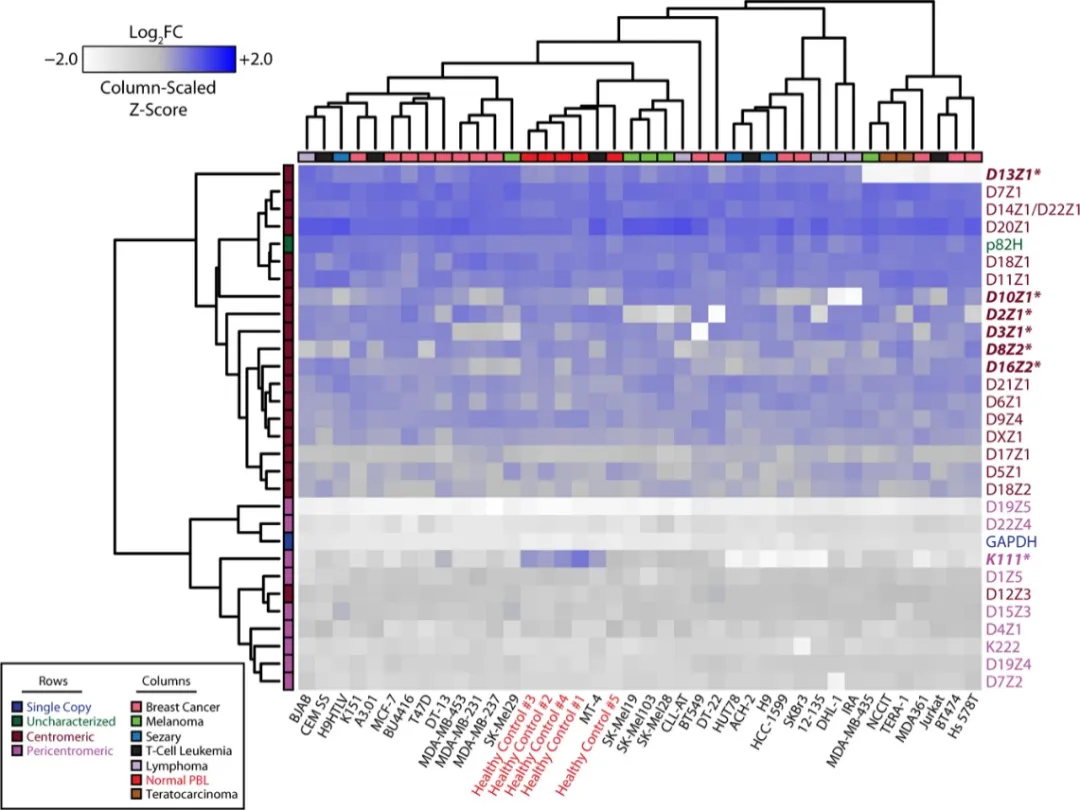
图8、 多种癌细胞系着丝粒DNA的异质性。热图表示通过qPCR从健康细胞核癌细胞获得的50ngDNA中的α着丝粒阵列丰度。标有星号表示与健康对照相比,各种癌细胞系具有显著变化。
# 04
总结
作为基因组的暗物质,过去20年着丝粒区域的基因组组装一直是一个难题,限制了我们对它们的认识。随着长读长测序技术的发展,T2T基因组变成现实。近期在对不同物种、个体的着丝粒序列的研究中,我们已经窥探到了着丝粒令人惊叹的高度可塑性,以及它们在核型演化、生殖隔离与物种形成、非整倍体疾病中的重要作用。
然而,还有很多谜题亟待解答,例如着丝粒如何在物种间多样演化?着丝粒的表观遗传调控与序列之间是如何互作?有哪些特征使细胞在分裂中容易出现染色体错误分配?着丝粒随生命周期、在癌细胞、衰老细胞和正常细胞之间如何变化等。
参考文献
[1]H. S. Malik and S. Henikoff, Major Evolutionary Transitions in Centromere Complexity, Cell, vol. 138, no. 6, pp. 1067–1082, Sep. 2009, doi: 10.1016/j.cell.2009.08.036.
[2] M. Naish and I. R. Henderson, The structure, function, and evolution of plant centromeres, Genome Res., vol. 34, no. 2, pp. 161–178, Feb. 2024, doi: 10.1101/gr.278409.123.
[3] C. Y. Y. Wong, Y. H. Ling, J. K. H. Mak, J. Zhu, and K. W. Y. Yuen, “Lessons from the extremes: Epigenetic and genetic regulation in point monocentromere and holocentromere establishment on artificial chromosomes,” Exp. Cell Res., vol. 390, no. 2, p. 111974, May 2020, doi: 10.1016/j.yexcr.2020.111974.
[4] U. Yadav, “Centromere,” in Encyclopedia of Animal Cognition and Behavior, J. Vonk and T. K. Shackelford, Eds., Cham: Springer International Publishing, 2022, pp. 1153–1157. doi: 10.1007/978-3-319-55065-7_13.
[5] H. Muller, J. Gil, and I. A. Drinnenberg, The Impact of Centromeres on Spatial Genome Architecture, Trends Genet., vol. 35, no. 8, pp. 565–578, Aug. 2019, doi: 10.1016/j.tig.2019.05.003.
[6] D. P. Melters et al., Comparative analysis of tandem repeats from hundreds of species reveals unique insights into centromere evolution, Genome Biol., vol. 14, no. 1, p. R10, Jan. 2013, doi: 10.1186/gb-2013-14-1-r10.
[7] T. Akera et al., Spindle asymmetry drives non-Mendelian chromosome segregation,Science, vol. 358, no. 6363, pp. 668–672, Nov. 2017, doi: 10.1126/science.aan0092.
[8] T. Kumon et al., Parallel pathways for recruiting effector proteins determine centromere drive and suppression,Cell, vol. 184, no. 19, pp. 4904-4918.e11, Sep. 2021, doi: 10.1016/j.cell.2021.07.037.
[9] F. K. Mastrorosa et al., Complete chromosome 21 centromere sequences from a Down syndrome family reveal size asymmetry and differences in kinetochore attachment, BioRxiv , Feb. 26, 2024. doi: 10.1101/2024.02.25.581464.
[10] A. K. Saha et al., The Genomic Landscape of Centromeres in Cancers, Sci. Rep., vol. 9, no. 1, p. 11259, Aug. 2019, doi: 10.1038/s41598-019-47757-6.
[11]V. Barra and D. Fachinetti, The dark side of centromeres: types, causes and consequences of structural abnormalities implicating centromeric DNA, Nat. Commun., vol. 9, no. 1, Art. no. 1, Oct. 2018, doi: 10.1038/s41467-018-06545-y.










