Nat Commun|冯少鸿团队揭示雀形目鸟类利用古病毒序列调控大脑基因
2024年4月12日,浙江大学生命演化研究中心和良渚实验室的冯少鸿团队发表了鸟类基因组中古老病毒来源的转座元件演化相关的研究成果。该研究基于万种鸟类基因组计划(Bird 10K Genomes project, B10K)第二阶段的数据,对鸟类基因组中的内源性病毒序列开展系统研究,分析了鸟类基因组整体上相对于其他脊椎动物拥有更少内源性病毒序列的潜在原因,发现鸟类有着更高水平清除不断扩张的病毒序列的演化压力。研究还发现一类内源性逆转录病毒K(ERVK)伴随着雀形目鸟类辐射性物种分化而在这些鸟类基因组中积累,其残留的序列可能作为顺式调控元件影响鸣禽大脑基因表达。该研究已经以“Adaptive expansion of ERVK solo-LTRs is associated with Passeriformes speciation events”为题,发表于Nature Communications杂志。

嵌入宿主基因组的古病毒
病毒与宿主在漫长的演化过程中既对抗又合作,对宿主产生深远影响。一些RNA病毒序列可以整合进宿主基因组,并具有转录活性,且在宿主基因组中不断扩张,这些病毒序列被称为内源性逆转录病毒(Endogenous retroviruses,ERVs)。逆转录病毒经由绑定、释放、逆转录与整合四个步骤,完成其在宿主基因组的内源化进程;当逆转录病毒侵染的宿主细胞为生殖细胞时,内源性逆转录病毒可以通过孟德尔遗传在代际之间进行垂直传播。内源性逆转录病毒在结构上为两端为高度相似的长末端重复序列(Long terminal repeats,LTRs),中间为病毒蛋白编码序列,也被称为LTRs转座子。由于ERVs具有转录活性,转录形成的RNA序列可以再次侵染宿主基因组,因此可以在宿主基因组中寄生并不断繁殖,产生大量的新拷贝。比如在人基因组中自身蛋白编码序列只占约2%,但内源性逆转录病毒序列(ERVs)却占比高达8%,近些年研究发现ERVs在癌症、衰老、细胞分化等多个重要生物学过程中发挥重要调控作用,对人类疾病和健康具有深远影响。
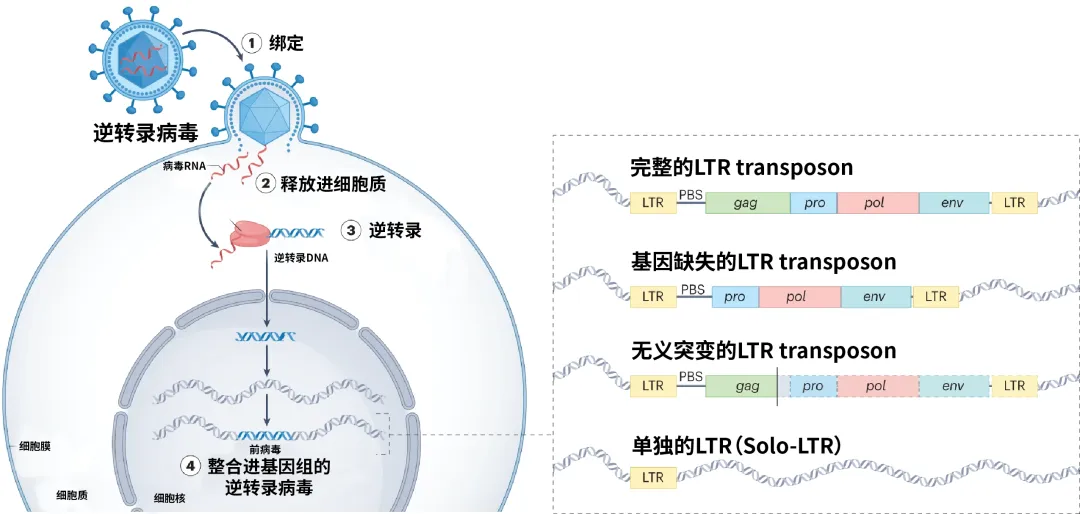
逆转录病毒的内源化过程:绑定、释放、逆转录与整合。(编辑自Dopkins and Nixon,2024)
由于遗传变异或者重组原因,大部分内源性逆转录病毒不再拥有完整的原始病毒的基因,在宿主基因组中可能以四种形式存在,即完整的LTR转座子、存在基因缺失的LTR转座子、存在无义突变的LTR转座子和单端残余的LTR序列(Solitary long terminal repeat,Solo-LTR)。其中单端LTR序列的形成主要是由于转座子两端高度相似的长末端重复序列会发生不平衡的同源重组,从而删除中间外源病毒序列,而仅在宿主基因组上留下Solo-LTRs的残留印记。宿主通过这种机制,可以清除掉其基因组中的内源性病毒序列。因此内源性病毒序列的扩增速度与Solo-LTRs的形成频率会极大地影响基因组的大小,在特殊情况下也可能会删除原本属于宿主基因组的序列。
鸟类受到更强的选择压力
清除内源性病毒序列
研究团队通过与爬行类、哺乳类等其他羊膜动物进行比较,揭示了鸟类基因组内形成Solo-LTRs的频率更高,即具备更高水平的清除内源性病毒序列的潜力,这一现象可能与鸟类相对于其他爬行类和哺乳类有更小基因组有关。研究团队推测如此高频形成Solo-LTRs的演化压力,可能与早先报道的较低的古病毒侵染频率,共同造成了鸟类内源性病毒元件(Endogenous viral element,EVEs)偏少的基因组格局。
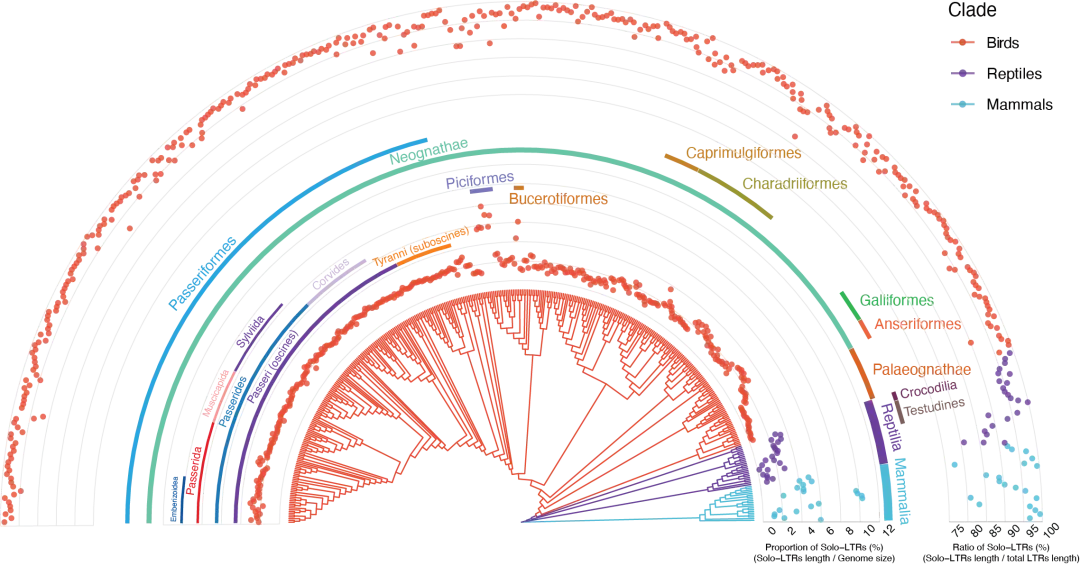
鸟类、爬行类和哺乳类Solo-LTRs基因组占比与形成频率图。
内圈表示Solo-LTRs相对于基因组大小的比例。外圈表示Solo-LTRs与总LTRs长度的比率,代表Solo-LTRs的形成频率(陈光霁绘)
ERVK Solo-LTRs伴随着
雀形目鸟类的物种分化而扩张
在现生鸟类1万多种鸟类中,约有6600多种鸟类为雀形目鸟类。雀形目鸟类起源可追溯至距今约5070万年前,并在距今约2240万年前发生了快速地辐射性演化事件,带来了绿森莺所属的莺小目(Sylviida)鸟类、穗鵖代表的鹟小目(Muscicapida)鸟类和斑胸草雀(珍珠鸟)所属的雀小目(Passerida)鸟类的物种多样性。雀形目的鸟类常被统称为“鸣禽(songbird)”,这是因为鸣禽亚目(Passeri)鸟类占据了绝大多数的雀形目鸟类多样性,他们也因鸣唱用的肌肉构造发达而善于鸣唱。鸣禽的发声学习(vocal learning)与人类的婴儿的习得语言(speech acquisitio)在行为和神经上有许多相似之处。
雀形目鸟类贡献现生鸟类超60%的物种多样性(编辑自:Feng, et al. 2020,陈星绘)
研究团队发现绝大部分的ERVs会被抑制在较低的水平,唯独内源性逆转录病毒K来源的Solo-LTRs序列在雀形目鸟类基因组中大量累积,而且其拷贝数随着物种分化事件的出现而逐渐增多。依据最新的鸟类生命之树,研究发现起源于ERVK的Solo-LTRs序列在雀形目鸟类中爆发性增长,特别在雀小目鸟类中,伴随着这一类群鸟类经历的物种分化事件拷贝数逐渐增多。研究团队推测ERVK在大约2240万年前开始非常活跃,伴随雀形目鸟类物种辐射性演化历程不断插入这些鸟类基因组中,而由于宿主同时有很强的清除能力,最终使得ERVK以Solo-LTRs形式残留在了雀形目鸟类基因组中。
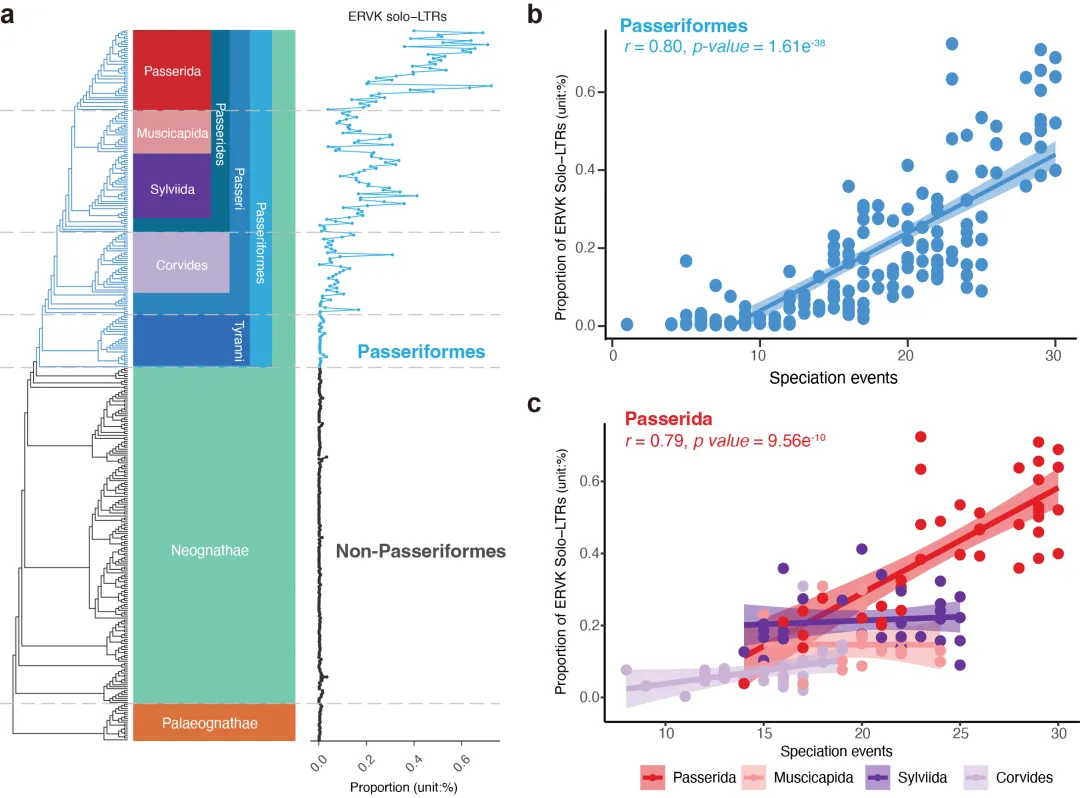
古老的内源性逆转录病毒K Solo-LTRs残留序列在雀形目鸟类中呈现不断累积的趋势,并与雀形目鸟类物种分化事件呈现正相关(陈光霁绘)
在漫长的演化历史中,受内源逆转录病毒K(ERVK)爆发性侵入宿主基因组不仅发生着雀形目鸟类中,在哺乳动物同样经历了辐射演化的类群如啮齿类和灵长类也曾经受了ERVK的大量入侵。在人类基因组中,ERVK的插入可以追溯至猿类灵长目的祖先(距今约5500万年前),其爆发事件可以追溯至大约2000万年前人猿总科和人类的祖先。早新近纪时期起源的鼠科啮齿类的基因组中也报道了相当高比例的ERVK序列。在多个类群中ERVK的共同爆发可能暗示着:ERVK的古病毒序列可能广泛分布于古大陆上,并在古近纪-新近纪界线期间(距今约2240万年前)频繁地整合到这些宿主基因组中。
古老病毒序列为宿主基因
调控提供材料
尽管这些序列元件是古病毒感染宿主留下的遗迹,绝大多数在鸟类基因组中已不再有完整的病毒基因功能,但它们却为鸟类基因组带来了调控功能上的遗传原材料。研究团队发现雀形目鸟类基因组中的多个基因可能受到了插入的ERVK Solo-LTRs序列的调控。如研究团队发现鸣禽斑胸草雀ITGA2基因的上游区域存在一段由ERVK Solo-LTRs插入带来的全新的调控区域,该调控区域可能受到具有激活信号的组蛋白第3亚基27号赖氨酸的乙酰化信号(H3K27ac)的影响。据报道,ITGA2基因的表达可能与另一鸣禽:白喉麻雀的鸣唱类型有关。
通过荧光原位杂交实验,研究团队发现潜在受到古老病毒的ERVK Solo-LTRs调控的ITGA2基因在斑胸草雀(鸣禽)鸣唱控制上游脑区呈现更高表达情况;对比在鸡基因组中ITGA2的同源基因并不存在这一调控元件,并未出现这一表达模式。进一步利用双荧光素酶报告基因实验,研究团队验证了该段插入序列具有的顺式调控活性。
研究团队推测,古老病毒的不断累积不仅为鸣禽鸟类提供了调控创新原材料,也促进了雀形目鸟类在适应性辐射演化进程中的遗传和性状多样性。这一发现为古病毒序列调控宿主基因功能从而影响宿主演化提供了一个例证。
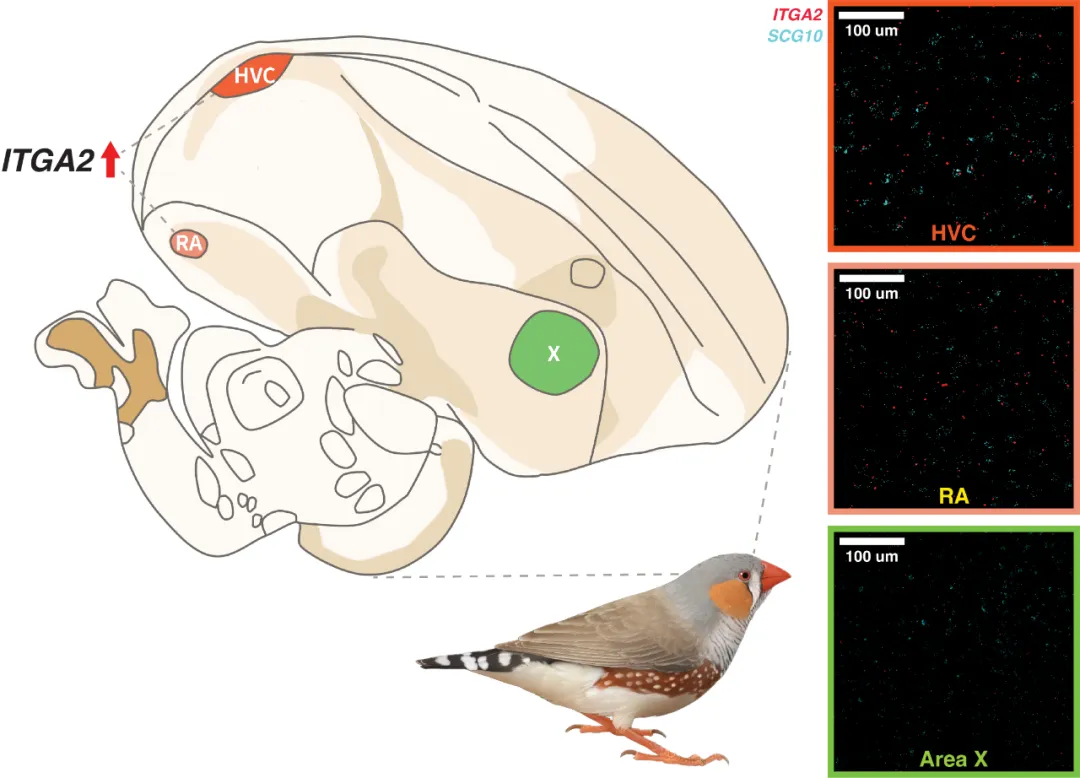
HVC、RA和X是鸣禽鸣唱控制通路的相关脑区,在斑胸草雀大脑的HVC区和RA区中都检测到了ITGA2基因的高表达(陈星、冉浩、杨宇与陈光霁等制图)
古老病毒序列可能仍在
不断改变当下鸟类基因组结构
尽管经历了相似的爆发性扩张事件,但ERVK在哺乳动物和鸟类的基因组中却走向了截然不同的演化路径。在哺乳动物中,尤其是在人类中,HERV-K仍然保留了编码病毒蛋白并形成病毒颗粒的能力。这些颗粒参与了宿主胚胎发育过程中的多个细胞过程,并与细胞衰老相关。
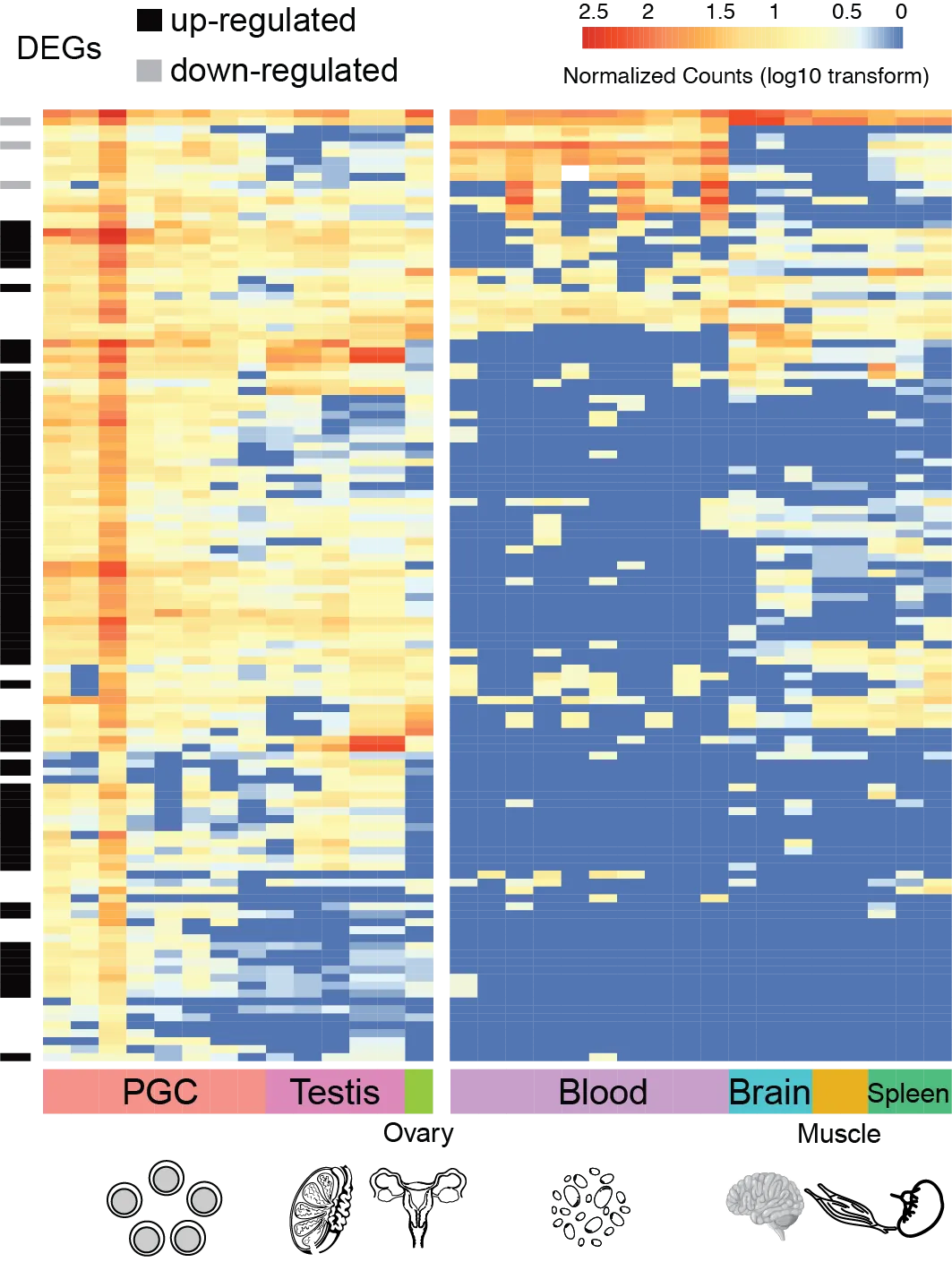
转录组学数据揭示内源性逆转录病毒K在斑胸草雀生殖相关组织中的高活性(陈光霁绘)
与此相反,在鸟类的演化过程中,可能面临着清除内源逆转录病毒(ERV)的选择压力,导致鸟类基因组内大量的ERVK病毒蛋白序列高频的形成Solo-LTRs。但,研究团队不仅在现生鸣禽斑胸草雀(珍珠鸟)野生群体重测序数据中,观察到内源性逆转录病毒K Solo-LTRs呈现基因组插入位点的多样性;在转录组数据中还发现在斑胸草雀生殖系统中(特别是原始生殖细胞中)内源性逆转录病毒K基因呈现出很高的表达活性。而在生殖系统中的高活性是古老病毒序列得以垂直传播的先决条件。
这些结果提示,古老的病毒序列来源的逆转录病毒K仍然活跃的存在于雀形目鸟类基因组中,直至今日,这些序列仍在不断地改变雀形目鸟类的基因组结构,影响鸟类的遗传多样性组成。
论文信息
论文标题:Adaptive expansion of ERVK solo-LTRs is associated with Passeriformes speciation events
上线时间:2024年4月12日
发表期刊:Nature Communications
DOI:10.1038/s41467-024-47501-3
作者署名:中国科学院大学与武汉华大生命科学研究院联合培养博士研究生陈光霁为文章第一作者。浙江大学生命演化研究中心和良渚实验室的冯少鸿研究员与复旦大学崔杰教授为文章的通讯作者。浙江大学生命演化研究中心张国捷教授、余丹、孙丹阳、卢妍林,中国科学院上海免疫与感染研究所李祥博士、王晓静博士,华侨大学硕士研究生杨宇、柯荣秦教授为文章共同作者。该项目获得了深圳国家基因库、良渚实验室、浙江大学长三角智慧绿洲创新中心提供的计算资源支持,以及国家自然科学基金与国家重点研发计划的大力支持。










