用基因组来观鸟,你会发现些什么?
地球上约有一万种鸟类,无论是在茂密的森林、广袤的海洋,还是炙热的沙漠、寒冷的极地,都能见到它们的身影。
有这么一群被称为观鸟者的人,他们扛着“长枪短炮”在自然中追逐着鸟儿的足迹,观察鸟儿的外形,倾听鸟儿的鸣唱,描绘鸟儿的行为。我们亦是观鸟者,但不同的是,我们并非通过镜片,而是透过基因组来解析鸟儿。
为此,我们集结了一批志同道合之士于2015年发起了万种鸟基因组学研究计划(https://b10k.genomics.cn/,即B10K,The Bird 10,000 Genomes Project)¹,目的是解读现生所有鸟类的基因组数据,完成基于全基因组水平鸟类生命之树的构建,鸟类遗传变异和性状差异关联的解码,分子演化、生物地理及物种多样性格局之间关系的解析等一系列重要的科学问题。
从2014年的48种鸟²,到2020年的363种鸟³,再到如今我们已经累计完成近3,000种鸟类组学数据的解读。
一路行来,借由基因组数据,我们都发现了什么呢?今日,就来浅谈一二吧。

图一
2014年12月12日,B10K目(Order)阶段研究成果在《科学》杂志上以专刊形式发表了八篇关于鸟类物种演化的文章。

图二
2020年11月12日,B10K科(Family)阶段首批研究成果在《自然》杂志上以封面文章的形式同期发表了两篇文章。

图三
基于48种鸟类全基因组序列构建的系统演化发育关系示意图,该成果重新确定了许多曾在历史上具有极大争议的鸟类的演化地位。Jon Fjeldså绘制。
鸟类基因组
与其他羊膜类动物相比,鸟类基因组不仅偏小,且物种间差异不大(鸟类0.9~2.1 Gb,哺乳动物1.6~6.3 Gb)⁴。这种现象通常被认为是鸟类适应飞行的结果。因为小的基因组有利于减少代谢消耗,鸟类基因组的大小随着与飞行有关的相对肌肉量的增加而减少⁵⁻⁸。例如,在已发表的鸟类中,基因组最小的蜂鸟拥有着最高超的飞行技术。在古颚类的平胸目和䳍形目鸟类基因组中,转座元件(transposable elements,TEs)不同程度的收缩和扩张对其基因组结构起到了很强的塑造作用,并且TEs的收缩速率在丧失了飞行能力的平胸目鸟类中要小于仍具备飞行能力的䳍形目鸟类⁹。
基因组研究表明,鸟类基因组的大小与TEs含量的减少、非编码区的减少、以及一些基因的丢失等因素有关2。在科(Family)水平上,96.1%的鸟类TEs的含量是低于15%的,即使是具有异常高TEs含量的绒啄木鸟和戴胜,也要远小于哺乳动物中高达35%-52%的TEs含量2,3。并且,由于内含子和基因间区的减少,基因在基因组上的分布也变得更加紧凑了2,6。
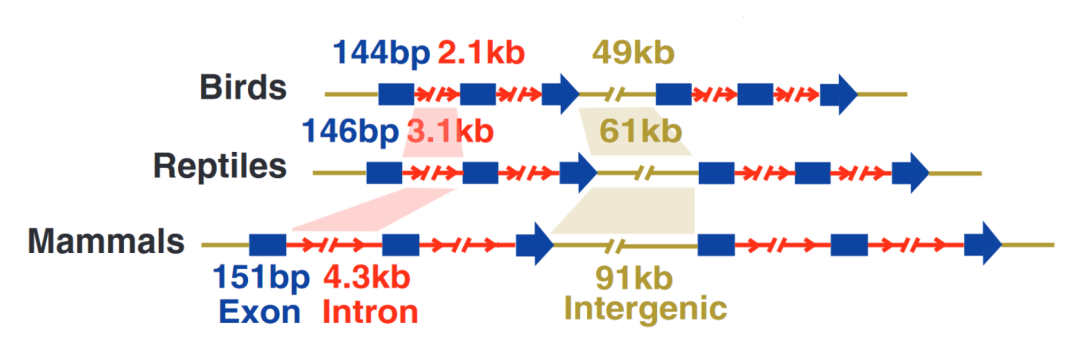
图四 鸟类、爬行动物和哺乳动物基因组内含子、外显子和基因间区域平均大小的比较。
与此同时,利用基因组数据,我们还鉴定到一些在祖先中存在,但在现存鸟类中已经丢失了的基因。比如,多个与牙釉质和牙本质形成相关的基因在所有鸟类的基因组中都已经丢失或者只剩下残留的序列,因此它们都丧失了生成牙齿的功能,成为了一群无“齿”之徒10。
为何鸟类物种内部基因组大小相对稳定?一些研究认为是由于鸟类演化过程中较少的DNA获取和丢失造成的11,12,而一些研究则更倾向于将维持鸟类基因组相对恒定大小的机制归功于通过转座子扩增获得DNA和通过缺失事件失去DNA之间的动态平衡4。
鸟类生命之树
达尔文将生命演化的历史比喻为一棵树,根部代表共同祖先,其上的枝叶是由共同祖先不断分叉演化形成的。重构鸟类生命之树一直是过去两个世纪中鸟类学家的主要目标。
如果我们说,在将现代鸟类分成古颚类(Palaeognathae,包括鸵鸟及其近亲)、鸡雁类(Galloanserae,包括鸡鸭等)和新鸟类(Neoaves,所有剩余约95%的鸟类)这三个主要大类的问题上基本不存在争议的话,那么,关于新鸟类内部主要分支之间的关系可谓是众说纷纭了。举个简单的例子,生活在亚马逊流域湿地林溪边的麝雉(Opisthocomus hoazin,拼音shè zhì),它的演化位置至今仍是一个谜团:在我们的研究中,麝雉的近亲是鹤和鸻鹬类;有的研究认为它们与包括蜂鸟等小型悬停鸟类在内的群体关系紧密;也有研究把它作为核心陆鸟类的外群。

图五 不丹邮票上的麝雉。麝雉雏鸟的翅膀上有两个坚硬的钩爪,拥有攀爬的技能点,长出飞羽之后就脱落了。麝雉的嗉囊极大,几乎占了体重的1/3,不仅能够储存食物也担负了一定的消化作用,就像牛的瘤胃一样。
事实上,这些充斥着大量矛盾冲突的新鸟类主要分支之间,往往有着非常短的内枝,意味着它们都是在非常短的时间内演化出现的13-15。我们知道,鸟类在约6500万年前的白垩纪末期生物大灭绝事件中幸存下来,继而迅速地演化出大量的新鸟类物种,经历了辐射性物种大爆发。而伴随着此类辐射性大爆发事件发生的不完全谱系分选事件(incomplete lineage sorting,ILS)正是造成新鸟类主要分支之间混乱关系的首要“元凶”13,16。
2014年,基于48只鸟的全基因组数据,我们构建了当时最具可信度的鸟类目 (Order)级别生命之树,重新确定了许多曾在历史上具有极大争议的鸟类的演化地位13。随后,Prum等人在2015年通过对更多的198个鸟类物种,但主要是基于部分保守的编码区序列也构建了鸟类系统发育关系17。这个物种树与我们在2014年发表的结果之间存在着颇多矛盾,并且集中在上文中提到的新鸟类主要分支之间。
随着越来越多的研究开始尝试解析鸟类生命之树,种种矛盾层出不穷。人们也关注于矛盾的可能来源,以我们和Prum等人的结果为例,矛盾的症结点在于构树时所使用的数据类型,而非物种抽样问题18,19。即,在使用编码区数据时更倾向于得到Prum等人的结果,而在使用非编码数据时则更支持2014年发表的结果。

图六 综合比较来自7个研究中的鸟类系统发育关系之间的矛盾20。每个分支节点上,颜色方块所在的位置对应不同的研究,如果是叉(×)则表示该研究不支持我们在2014年发表的结果。
鸟类物种中存在着许多像麝雉这样的“trouble makers”,想要彻底绘制出鸟类生命之树,依然是充满挑战的。怎样实现更高的物种覆盖度,构建更优的比对序列,获得更齐全的多维度数据,树状结构是否足以来勾勒物种演化进程……这都是等待我们来解决的有趣谜题。
纵观古今,鸟类研究的历史由来已久。从我国最早的鸟类志《禽经》中“股有彩纹曰锦鸡”,到达尔文观察笔记中的加拉帕戈斯群岛雀科鸣禽,博物馆中珍贵的鸟类化石,各种形态、生理、行为的记录和研究,再加上古DNA、组学等技术手段的加持,我们从鸟类身上学到的知识还有很多很多。欲知详情,请听下回分解!
参考文献
1. Zhang, G. et al. Genomics: Bird sequencing project takes off. Nature 522, 34, (2015).
2. Zhang, G. et al. Comparative genomics reveals insights into avian genome evolution and adaptation. Science 346, 1311-1320, (2014).
3. Feng, S. et al. Dense sampling of bird diversity increases power of comparative genomics. Nature 587, 252-257, (2020).
4. Kapusta, A., Suh, A. & Feschotte, C. Dynamics of genome size evolution in birds and mammals. Proc Natl Acad Sci U S A 114, E1460-E1469, (2017).
5. Hughes, A. L. & Hughes, M. K. Small genomes for better flyers. Nature 377, 391, (1995).
6. Organ, C. L., Shedlock, A. M., Meade, A., Pagel, M. & Edwards, S. V. Origin of avian genome size and structure in non-avian dinosaurs. Nature 446, 180-184, (2007).
7. Wright, N. A., Gregory, T. R. & Witt, C. C. Metabolic 'engines' of flight drive genome size reduction in birds. Proc Biol Sci 281, 20132780, (2014).
8. Ji, Y. & DeWoody, J. A. Relationships Among Powered Flight, Metabolic Rate, Body Mass, Genome Size, and the Retrotransposon Complement of Volant Birds. Evolutionary Biology 44, 261-272, (2017).
9. Wang, Z. J., Chen, G. J., Zhang, G. J. & Zhou, Q. Dynamic evolution of transposable elements, demographic history, and gene content of paleognathous birds. Zool Res 42, 51-61, (2021).
10. Meredith, R. W., Zhang, G., Gilbert, M. T., Jarvis, E. D. & Springer, M. S. Evidence for a single loss of mineralized teeth in the common avian ancestor. Science 346, 1254390,(2014).
11. Shedlock, A. M. Phylogenomic investigation of CR1 LINE diversity in reptiles. Syst Biol 55, 902-911, (2006).
12. Janes, D. E., Organ, C. L., Fujita, M. K., Shedlock, A. M. & Edwards, S. V. Genome evolution in Reptilia, the sister group of mammals. Annu Rev Genomics Hum Genet 11, 239-264, (2010).
13. Jarvis, E. D. et al. Whole-genome analyses resolve early branches in the tree of life of modern birds. Science 346, 1320-1331, (2014).
14. Hackett, S. J. et al. A Phylogenomic Study of Birds Reveals Their Evolutionary History. Science 320, 1763-1768, (2008).
15. Claramunt, S. & Cracraft, J. A new time tree reveals Earth history's imprint on the evolution of modern birds. Sci Adv 1, e1501005, (2015).
16. Suh, A., Smeds, L. & Ellegren, H. The Dynamics of Incomplete Lineage Sorting across the Ancient Adaptive Radiation of Neoavian Birds. PLoS Biol 13, e1002224, (2015).
17. Prum, R. O. et al. A comprehensive phylogeny of birds (Aves) using targeted next-generation DNA sequencing. Nature 526, 569-573, (2015).
18. Reddy, S., Kimball, R. T., Pandey, A., Hosner, P. A. & Braun, E. L. Why Do Phylogenomic Data Sets Yield Conflicting Trees? Data Type Influences the Avian Tree of Life more than Taxon Sampling. Systematic Biology 66 (2017).
19. Braun, E. L. & Kimball, R. T. Data Types and the Phylogeny of Neoaves. Birds 2, 1-22, (2021).
20. Bravo, G. A., Schmitt, C. J. & Edwards, S. V. What Have We Learned from the First 500 Avian Genomes? Annual Review of Ecology, Evolution, and Systematics 52, 611-639, (2021).










