Cell Reports:李启业/张国捷团队揭示动物界RNA编辑机制的起源和演化过程
RNA编辑是指RNA分子上发生碱基插入、删除或替换,从而导致成熟的RNA与最初编码它的DNA模版产生差异的一种基因转录后调控机制。RNA编辑系统在真核生命演化史上独立起源了多次,目前在原生生物、后生动物、植物、真菌等不同类群中均有报道,被认为在RNA成熟和基因转录后调控过程中起着重要作用1。但这其中,后生动物的RNA编辑系统又最为特别。在其他真核类群中,RNA编辑的底物主要是tRNA或细胞器转录的mRNA,影响范围有限;而后生动物的RNA编辑系统能大量修改细胞核转录出来的mRNA。在章鱼等头足类动物中,甚至有高达50%的核基因会发生转录后的RNA编辑2。这提示人们在研究动物的基因产物时,不能完全相信DNA模版。
在动物界,最常见的RNA编辑现象是由ADAR酶介导的腺苷(A)到肌苷(I)的编辑。由于肌苷的化学性质跟鸟苷(G)相似,A-to-I编辑等价于在RNA序列中引进A-to-G的碱基替换,如发生在基因的蛋白编码区则有可能改变氨基酸的编码3。A-to-I编辑对动物的生长发育和环境适应等过程均有重要调控作用,其失调在人类中会导致神经系统疾病、免疫疾病以及癌症的发生4-6。然而,目前人们对这一重要转录后调控机制在动物界何时起源以及如何演化依然所知甚少。
2023年2月14日,华大生命科学研究院李启业与浙江大学生命演化研究中心张国捷教授联合其他国内外合作者在Cell Reports杂志在线发表题为On the origin and evolution of RNA editing in metazoans的研究论文,揭示了ADAR介导的RNA编辑机制是后生动物祖先的重要演化创新,并提出ADAR编辑系统最初的功能很可能是用于防止基因组重复序列衍生的内源双链RNA的积累,但又因其能诱发点突变的特性,可为自然选择提供更多遗传素材,因而在漫长的动物演化过程中被频繁招募到其他基因调控网络从而影响众多生物过程。
在本研究中,研究人员精心挑选了22个能代表动物不同演化阶段的物种,样品覆盖了后生动物的单细胞近亲(如领鞭毛虫)、较为原始的非两侧对称动物(如海绵、栉水母、丝盘虫)以及较为复杂的两侧对称动物(如昆虫、软体动物、脊椎动物)。研究人员对每个物种都进行了高深度的基因组重测序和链特异性转录组测序,并利用团队自行研发的RES-Scanner软件包7构建了这些物种的RNA编辑图谱(也即所有RNA编辑事件的集合)。
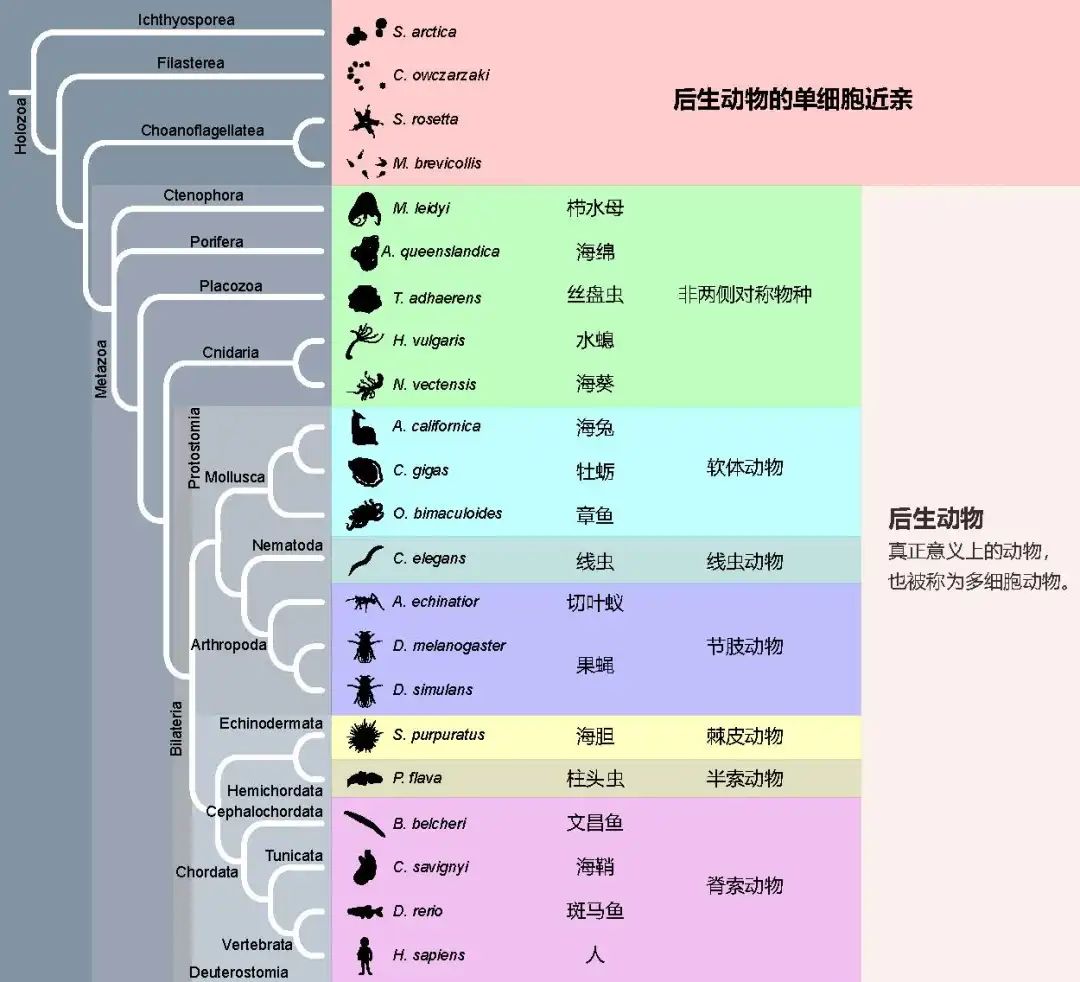
图1.本研究所涉及的22个物种
研究人员在除丝盘虫(一种扁盘动物)外的所有后生动物中均能识别到大量的RNA编辑事件,且90%以上都表现为A-to-G的碱基替换。与此同时,研究人员在这些物种的基因组中都能识别到至少一个ADAR基因。而在所有近缘的单细胞外群物种中,既不存在可靠的RNA编辑事件也不存在ADAR基因。这有力地说明了ADAR介导的RNA编辑机制起源于现存所有后生动物的共同祖先,是后生动物的一种演化创新。而这次古老的创新在随后长达8亿多年的动物演化史中被绝大多数动物门类保留下来,又进一步说明了这一机制对动物的重要性。
过去一度认为RNA编辑的主要作用类似于RNA可变剪切,能拓展基因的编码能力,使一个基因能产生更多的RNA和蛋白变体。然而,研究人员发现,在从海绵到人的绝大数后生动物中,A-to-I编辑都主要发生在基因组重复序列所转录的非编码RNA上,特别是年轻的重复序列,而发生在蛋白编码区的编辑事件非常稀少。相邻且反向的重复序列能互补配对形成长的双链RNA,这是ADAR酶作用的理想底物3。但值得注意的是,细胞内双链RNA的积累同时也是RNA病毒入侵的标志。此前在哺乳动物中就有研究发现,敲除ADAR酶会导致细胞内的双链RNA感应器(如RIG-I-like receptors,RLRs)错误把内源的双链RNA识别为外源的病毒,从而引起自身免疫反应8。有趣的是,RLRs也是后生动物的演化创新。研究人员还发现,演化过程中独立丢失了ADAR编辑机制的丝盘虫,其基因组中也同时丢失了识别双链RNA的RLRs。这些结果提示ADAR编辑机制的原始功能很可能是防止内源双链RNA的积累从而引起错误的免疫反应。研究人员还提出,双链RNA感受器的存在很可能是现存后生动物ADAR编辑机制被保留的主要自然选择压。
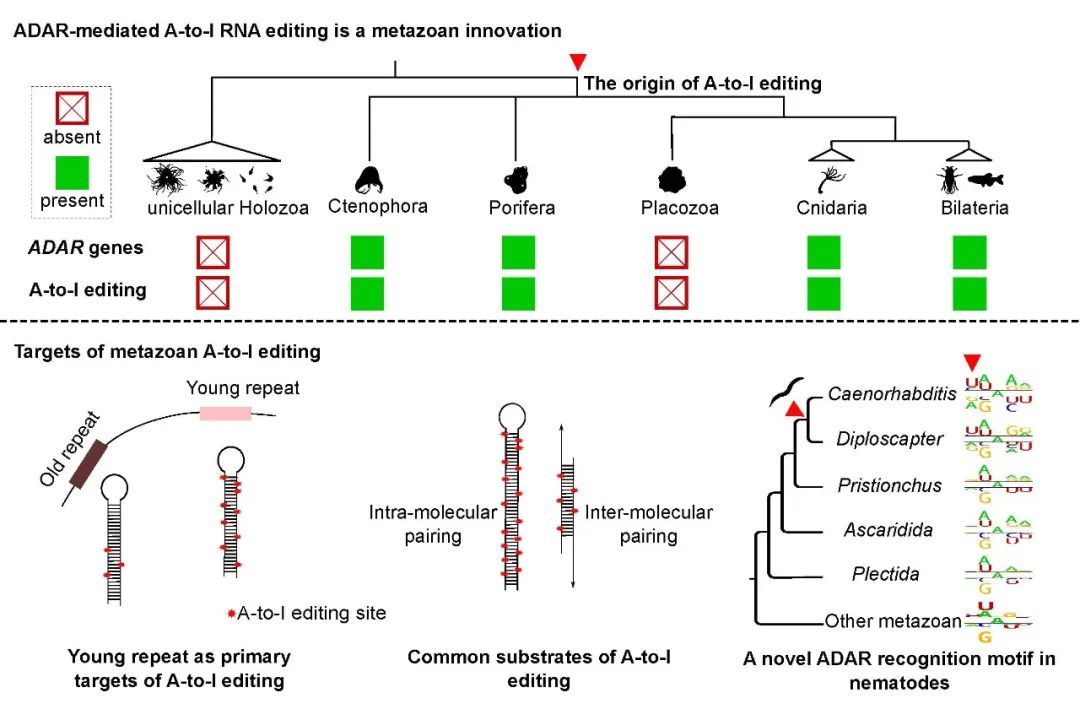
图2.ADAR介导的RNA编辑机制的起源和主要作用底物
RNA编辑最早被人熟知的功能是引起密码子的改变进而改变氨基酸编码(俗称recoding编辑),该功能已被广泛报道对神经传递相关蛋白有重要调节作用9。研究人员发现,recoding编辑现象在栉水母、海绵等早期分枝的后生动物中已经存在。然而让研究人员感到意外的是,除神经传递外,细胞骨架系统(包括其中的结构蛋白和动力蛋白)在更大范围的物种类群中发生了recoding现象,提示RNA编辑对于细胞骨架系统的功能具有广泛的调节作用,值得未来研究的关注。此外,研究人员还发现了天然反义转录本与其正义链基因转录本的配对,也是诱导A-to-I编辑的重要机制,在个别物种如海鞘中甚至是主要机制。然而,该机制在过去并不被重视10。
最后,研究人员还发现,尽管来自不同门类的动物已经分歧了数亿年,但不同物种中ADAR酶的识别motif基本保持一致,仅仅在秀丽隐杆线虫及其近亲物种中发生了变化。这可能对于正在研发利用物种内源ADAR酶进行位点特异RNA编辑技术的研究者来说是个好消息,因为他们目前基于哺乳动物开发的方法有望能快速应用到其他物种中。而通过motif改变的线虫与其近缘物种的比较,也让研究人员在ADAR酶上定位到了2个对ADAR酶motif识别可能起关键作用的氨基酸位点。
华大生命科学研究院李启业副研究员、浙江大学生命演化研究中心张国捷教授为该论文共同通讯作者。华大生命科学研究院张霈、朱元镇与中国科学院大学硕士研究生郭群飞为共同第一作者。该研究主要得到了国家自然科学基金的资助。
参考文献
1.Gray, M.W. (2012). Evolutionary origin of RNA editing. Biochemistry 51, 5235-5242. 10.1021/bi300419r.
2.Liscovitch-Brauer, N., Alon, S., Porath, H.T., Elstein, B., Unger, R., Ziv, T., Admon, A., Levanon, E.Y., Rosenthal, J.J.C., and Eisenberg, E. (2017). Trade-off between Transcriptome Plasticity and Genome Evolution in Cephalopods. Cell 169, 191-202 e111. 10.1016/j.cell.2017.03.025.
3.Nishikura, K. (2016). A-to-I editing of coding and non-coding RNAs by ADARs. Nat Rev Mol Cell Biol 17, 83-96. 10.1038/nrm.2015.4.
4.Zipeto, M.A., Jiang, Q., Melese, E., and Jamieson, C.H. (2015). RNA rewriting, recoding, and rewiring in human disease. Trends Mol Med 21, 549-559. 10.1016/j.molmed.2015.07.001.
5.Ben-Aroya, S., and Levanon, E.Y. (2018). A-to-I RNA Editing: An Overlooked Source of Cancer Mutations. Cancer Cell 33, 789-790. 10.1016/j.ccell.2018.04.006.
6.Rice, G.I., Kasher, P.R., Forte, G.M., Mannion, N.M., Greenwood, S.M., Szynkiewicz, M., Dickerson, J.E., Bhaskar, S.S., Zampini, M., Briggs, T.A., et al. (2012). Mutations in ADAR1 cause Aicardi-Goutieres syndrome associated with a type I interferon signature. Nat Genet 44, 1243-1248. 10.1038/ng.2414.
7.Wang, Z., Lian, J., Li, Q., Zhang, P., Zhou, Y., Zhan, X., and Zhang, G. (2016). RES-Scanner: a software package for genome-wide identification of RNA-editing sites. Gigascience 5, 37. 10.1186/s13742-016-0143-4.
8.Liddicoat, B.J., Piskol, R., Chalk, A.M., Ramaswami, G., Higuchi, M., Hartner, J.C., Li, J.B., Seeburg, P.H., and Walkley, C.R. (2015). RNA editing by ADAR1 prevents MDA5 sensing of endogenous dsRNA as nonself. Science 349, 1115-1120. 10.1126/science.aac7049.
9.Behm, M., and Ohman, M. (2016). RNA Editing: A Contributor to Neuronal Dynamics in the Mammalian Brain. Trends in genetics : TIG 32, 165-175. 10.1016/j.tig.2015.12.005.
10.Neeman, Y., Dahary, D., Levanon, E.Y., Sorek, R., and Eisenberg, E. (2005). Is there any sense in antisense editing? Trends in genetics : TIG 21, 544-547. 10.1016/j.tig.2005.08.005.










