靠晒太阳活下去的终极生存指南:质体内共生开启“无限续航”!
一、内共生事件——真核细胞复杂性演化的主要驱动力
约40亿年前,地球的生命形式简单而原始,生命依赖于环境中有限的资源生存。那时,大气中氧气含量极低,缺乏臭氧层保护,强烈的紫外线辐射不利于生命繁衍。约24-26亿年前,远古蓝细菌演化出光合作用的能力,开始大量释放氧气,逐渐改变了地球大气环境1。约15-20亿年前,真核生物祖先通过先后吞噬整合α-变形细菌和蓝细菌,分别获得了线粒体2和质体(叶绿体)3。这两次关键的内共生事件,赋予了真核生物高效的能量代谢和光合作用能力,推动了生物多样性和复杂生态系统的形成,是地球生命演化史上的重大里程碑。
“真核生物起源于内共生事件”这一简单语句蕴含深远意义。1995年,马古利斯(Lynn Margulis)和萨根(Carl Sagan)提出,生命通过协作而非单纯的竞争占领了全球4。然而,至今我们对两个原本独立的生命体如何融合成为新的生命形式仍知之甚少,这一过程可能涉及一系列复杂的机制,值得探讨。
二、探索内共生演化历程的挑战
尽管内共生理论已广为接受,但完全解开其复杂演化过程仍面临巨大挑战。由于永久内共生关系的建立发生在久远的历史时期,其形成过程的线索大多已淹没在漫长的演化历程中。线粒体起源于所有已知真核生物多样化之前,因此追溯其起源尤为困难。然而,与线粒体不同的是,质体除了最初的内共生事件外,还经历了多次次级和三级内共生事件5【图一】,在这些事件中,已经拥有质体的真核细胞被另一个真核细胞吞噬,并形成了新的共生关系。这种层层叠加的共生关系又加剧了光合自养生物谱系的复杂性及追溯其内共生演化历程的挑战。

图一 具有质体的真核生物谱系间演化关系(图片来源于引文5)。具有两层膜的初级质体存在于绿藻、红藻和灰胞藻中,具有三层/四层膜的次生质体存在于隐藻,定鞭藻,裸藻与 Stramenopile-Alveolate- Rhizarian 等门类中,其中分支的颜色表明该谱系的质体是来自绿藻还是红藻。
三、盗质体(Kleptoplasty)现象--从短暂内共生向永久内共生过渡
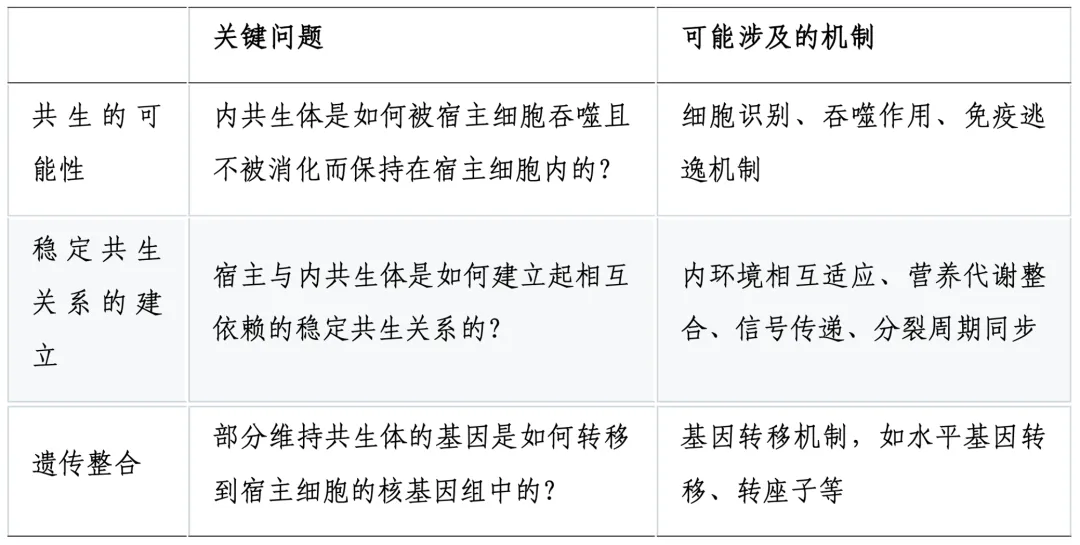
尽管质体在与其原始宿主细胞的长久协作中逐步地丧失了独立生存的能力,但至今我们仍发现质体可以被短暂并具有功能性地保留在与光合自养谱系距离甚远的异养生物细胞内【图二A】。例如,一些海洋软体动物(海蜗牛)能从其藻类食物中摄取质体,并将这些源自藻类的质体保存于其自身消化道细胞内,从而临时获得这些质体的光合自养能力。学界将这种现象称为盗质体现象。这些未被消化的质体结构与功能保持完整,被称为盗质体,能够短暂或长期进行光合作用(有时甚至贯穿新宿主的整个生命周期)【图二B】。类似的盗质体现象已在多个门类中被报道【图三】,包括鞭毛藻(dinoflagellates)、纤毛虫(ciliates)、盖孢虫(Katablepharid)、有孔虫(foraminiferans)和后生动物(sea slugs and marine flatworms)6。这种现象并不涉及质体的遗传整合,即这些质体不可增殖且没有被永久地整合进宿主的细胞结构中,因此不能通过遗传方式传递给后代。然而,这类现象可能代表了内共生演化的过渡阶段,为我们提供了一个独特的视角,观察两个原本独立的生命体如何从短暂的共生关系向持久性的内共生关系演化。
图二 质体跨界细胞内共生示例与功能演变。(A)盗质体在纤毛虫(左)和海蜗牛(右)细胞内分布;(B)维持非功能性、短期功能性与长期功能性盗质体的海蜗牛演化关系。(图片来源于引文7)。
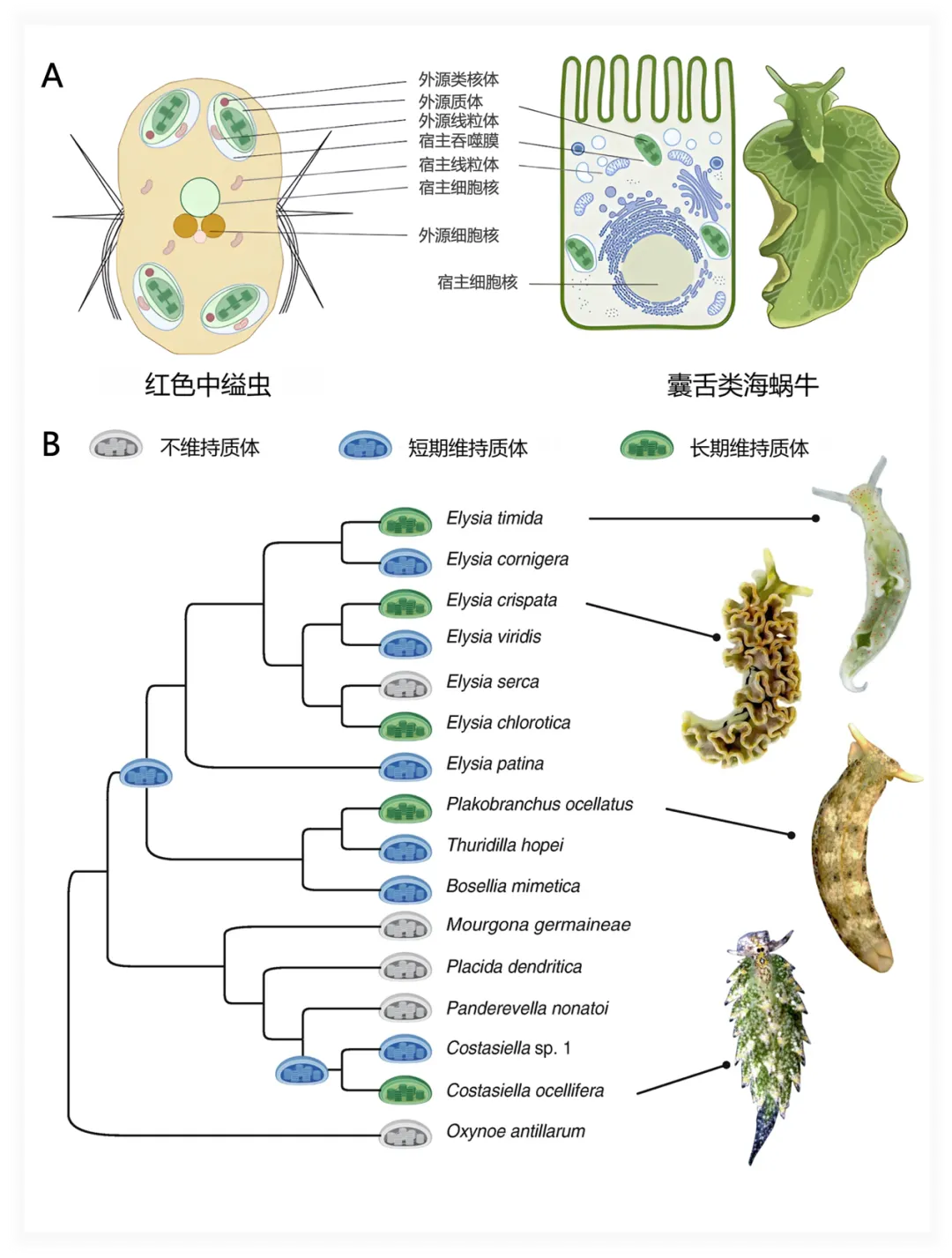
图三 质体在真核生物界的分布:永久性质体(实心圆圈)和盗质体与光合共生体(空心圆圈)在不同真核生物谱系间的分布与演化关系(图片来源于引文6)。
四、盗质体内共生系统演化的多样性与复杂性
宿主对盗质体的维持机制是一个复杂且多样化的过程,不同宿主采取的策略各不相同。在具有盗质体能力的海蜗牛细胞中,功能性质体可以在不依赖被摄取的藻核基因的情况下正常运作,最长可达数月。目前有研究显示海蜗牛基因组内暂未发现藻类来源水平基因转移或质体内共生基因转移现象8。这引发了一个重要的问题:海蜗牛如何在没有藻类核基因的情况下,给予盗质体能量和物质代谢基础,并为其提供抗氧化机制以抵御光氧化损伤?宿主与质体间建立蛋白质转运和代谢调控的整合,是建立稳定内共生关系的关键9。此外,被宿主捕获的质体可能获得额外包膜,导致质体膜数量增加,核编码质体靶向蛋白的转运机制更复杂。这些问题都尚待研究。
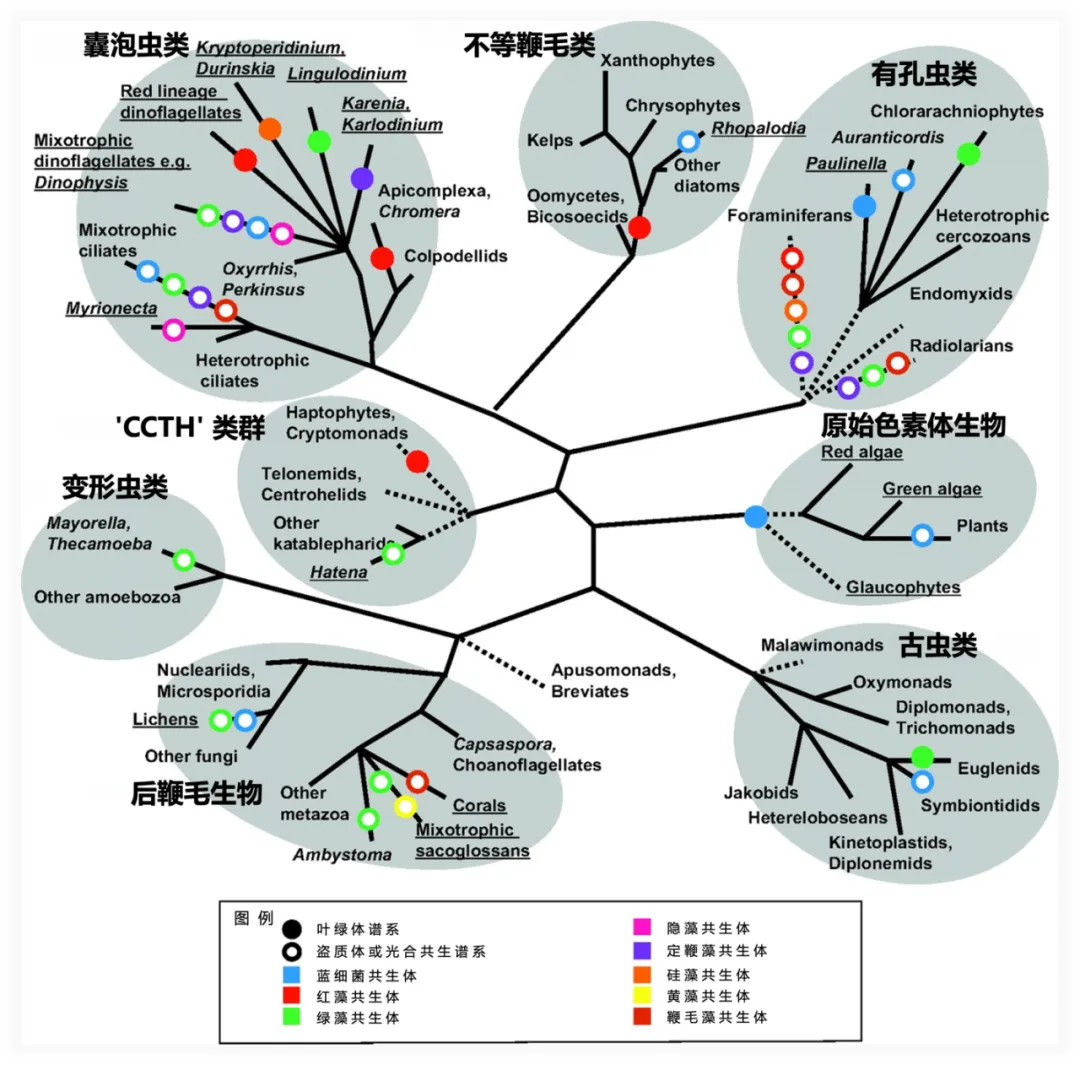
另一方面,一些宿主,例如纤毛虫Mesodinium rubrum【图二A左】,会同时维持其藻类食物的质体和细胞核。通过对M. rubrum 及其隐藻食物T. amphioxeia组成的盗质体系统进行转录组测序分析,研究人员发现其保留的藻来源的核DNA复制和代谢相关基因转录显著上调,而盗质体失去了光敏感性10【图四】。在与纤毛虫演化距离很远的鞭毛藻Nusuttodinium aeruginosum(其食物隐藻为Chroomonas sp.,宿主也同时维持藻类的质体与核)中,研究者也发现了同样的调控趋势11。这反映了在建立内共生关系初始阶段宿主对内共生体的整合与调控模式,并表明宿主对内共生体的控制有限。
图四 该模式图展示了与自由生活的隐藻Teleaulax amphioxeia基因转录模式相比,在纤毛虫宿主Mesodinium rubrum细胞内环境下,T. amphioxeia光依赖性基因转录变化消失,且即使在无光照条件下依然维持高表达水平。
五、宿主对质体的控制是永久内共生关系形成的关键
为了更好地理解这种复杂的机制,我们需要关注宿主对质体的控制是如何在长期的内共生关系中建立的。随着时间流逝,质体基因组在宿主细胞内环境中经历基因丢失,部分必需的质体基因或者藻类核基因转移到宿主核基因组上,在宿主细胞中表达并转运回质体。这种基因转移使宿主有机会获得新功能的基因并减少质体自身能量消耗。研究发现,在dinoflagellate和euglenozoan两大类群下的不同盗质体系统中均存在基因转移现象,且宿主核基因组中包含部分与盗质体不同谱系的质体靶向基因12,13。此外,大量测序数据表明,在部分具有永久质体的真核谱系中,质体的系统发生关系与宿主核基因组关系存在冲突。尤其是在含次级质体的藻类谱系中,其核基因组也包含大量并非来自其质体同一谱系的质体靶向基因14-18。
这提示了宿主对质体的控制可能是渐进式的(详见【图五】“shopping-bag” 模型19,20),也即宿主在与内共生质体完全整合前可能形成过一系列短暂的内共生关系,逐步通过基因转移整合质体靶向基因,获得建立内共生关系所需的预适应性。随着宿主对内共生质体的控制增强,质体的保留期可能会逐步增长,最终导致宿主和质体的遗传整合,宿主完全控制质体的生长和分裂。这一模式是否具有普遍性,还需要更多研究来证实。
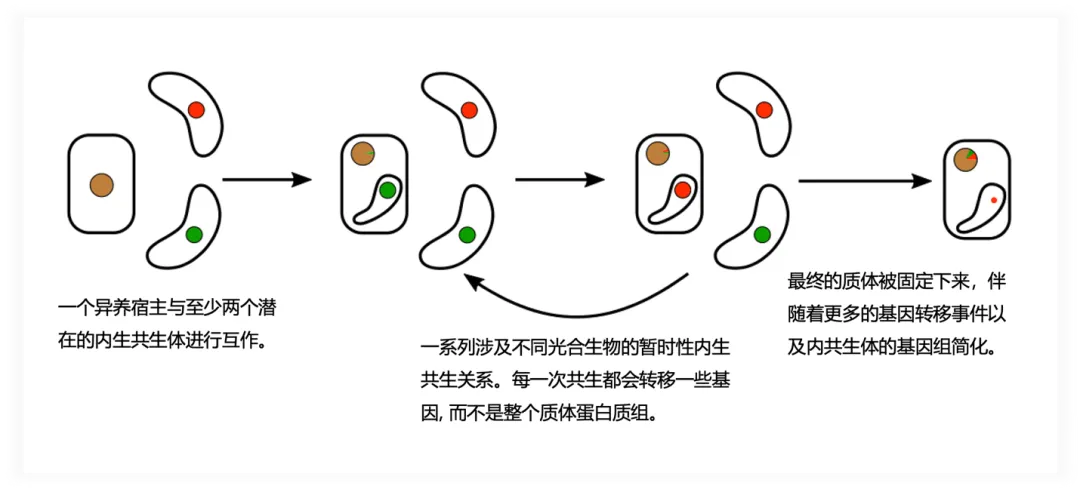
图五 “shopping-bag” 模型(图片来源于引文21)。
六、从生物协作到生物技术创新
质体跨界内共生揭示了生物演化的复杂性。从生态适应到基因组整合,每个层面都展现出生物的灵活性和创新性。这种现象不仅深化了我们对共生演化的理解,也为探索生命系统的适应性和可塑性提供了重要线索,并为生物技术应用提供了新思路,例如开发新型光合生物反应器。值得关注的是,近年来浙江大学医学院邵逸夫医院林贤丰、范顺武与浙江大学化学系唐睿康团队合作成功将植物类囊体(质体中的单层膜囊状结构)移植到哺乳动物细胞内,以改善退变损伤细胞的代谢功能障碍,这为治疗退行性疾病提供了新方法22。
在生物医学领域,细胞的代谢功能对健康至关重要。退行性疾病如骨关节炎可导致细胞代谢紊乱,限制能量原料代谢,进而导致细胞功能下降,加重疾病进展。为了解决这一问题,研究团队开发了 CM-NTUs(细胞膜封装纳米类囊体)系统,从菠菜中提取能量供应细胞器,作为“生物电池”补充动物细胞能量,并通过细胞膜封装以避免免疫排斥。该系统在光照下能自我合成能量原料,改善细胞代谢功能。
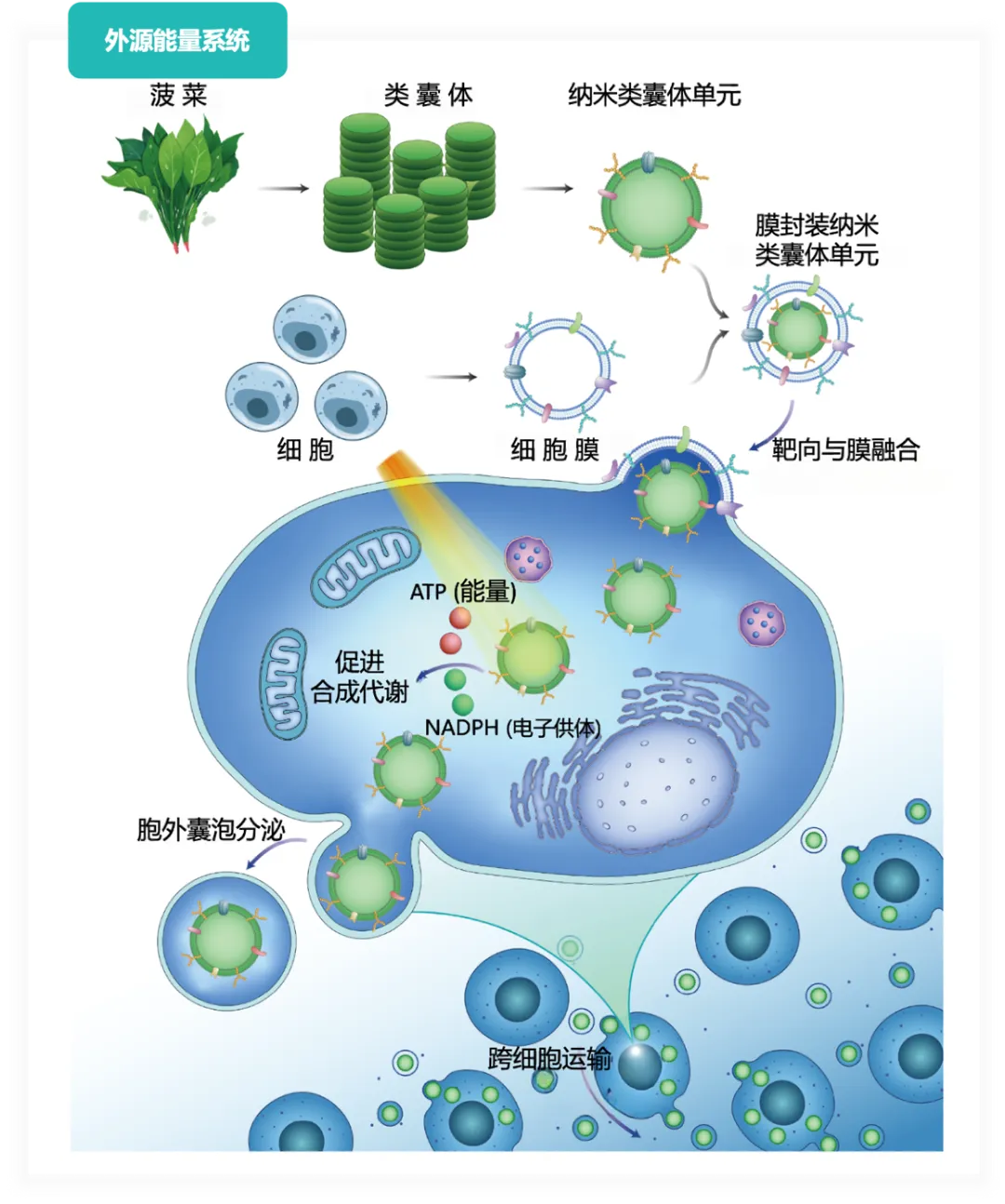
图六 CM-NTUs(细胞膜封装纳米类囊体)系统(图片由林贤丰团队提供)。
通过将植物类囊体移植到哺乳动物细胞内,CM-NTUs 系统能够有效提升细胞的合成代谢能力。在小鼠骨关节炎模型实验中,CM-NTUs 显示出良好的细胞渗透性和生物相容性,光照条件下增加了 ATP 和 NADPH 的产生,能够恢复退化的软骨细胞的代谢平衡。在术后 12 周的CM-NTUs光照治疗下,小鼠表现出软骨下骨板形态变化和骨赘形成减少【图七】。据研究人员描述,“原本相当于人类60岁状态的衰老退变软骨在几周内就恢复到20岁水平”。这一发现为再生医学中的细胞代谢调节提供了新的方向。
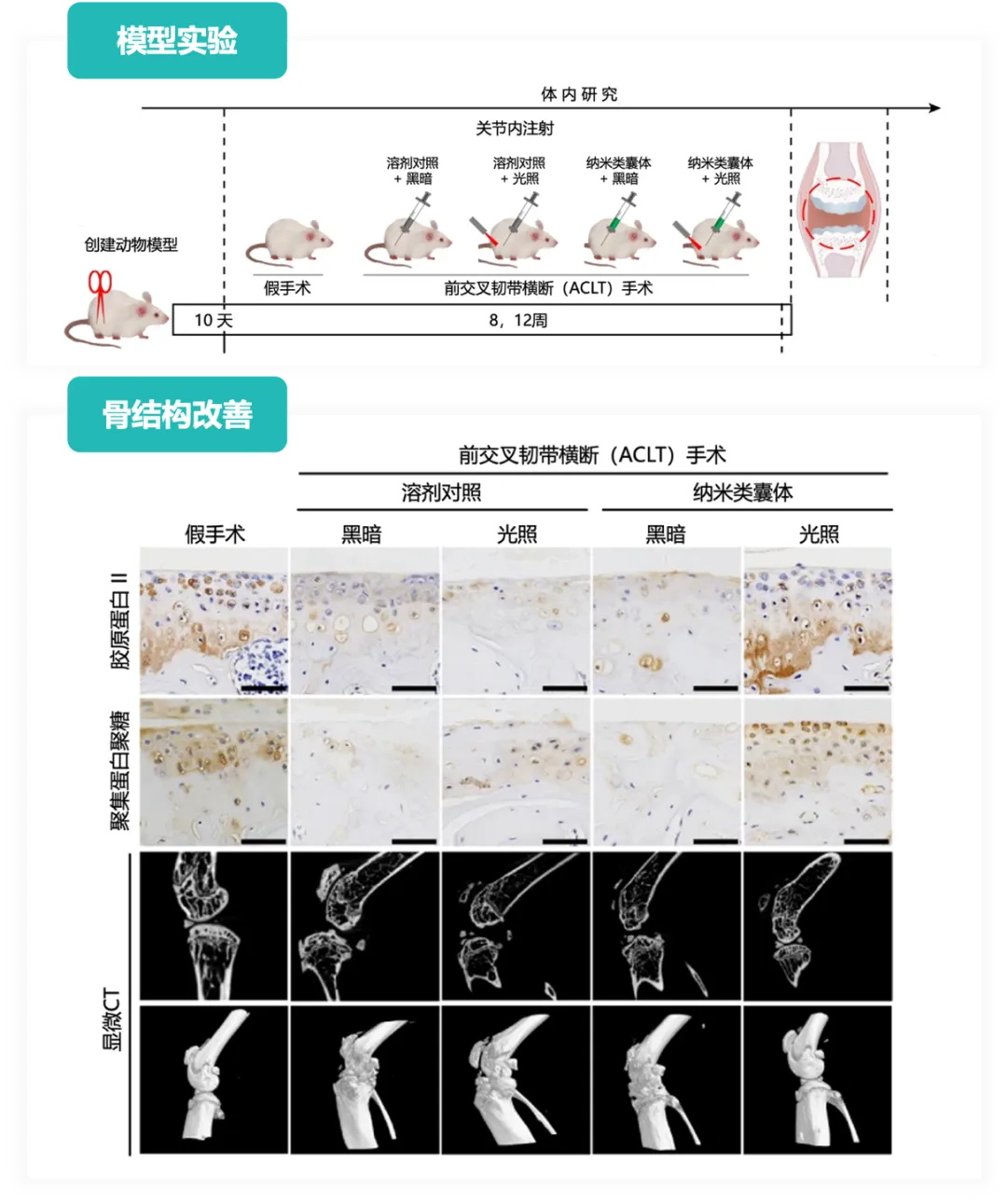
图七 纳米类囊体治疗对小鼠骨关节炎模型的体内影响(图片由林贤丰团队提供)。通过 ACLT 手术在小鼠中建立骨关节炎模型,并将小鼠随机分为接受CM-NTUs(细胞膜封装纳米类囊泡)治疗组和对照组。通过胶原蛋白II(Col II)和聚集蛋白聚糖的免疫组织化学染色结果,同时还包括膝关节的显微CT图像和三维图像,在第12周的纳米类囊体+光照治疗组中观察到骨结构改善。
研究人员还发现,通过调整光照的强度和时间,可以进一步优化 CM-NTUs 的代谢效果,为未来的研究提供了广阔的空间。深入研究盗质体系统,特别是在内共生体的识别与选择性、胞内物质运输和光合系统的长期维持等方面,将为这类应用研究提供新的见解和启发。尤其是在探索该系统在其他类型细胞中的疗效时,盗质体系统的研究显得尤为重要。这将有助于推动该系统在心脑血管的再生修复、肌肉萎缩等更广泛领域的应用。
参考文献
1. Shih, P. M., Hemp, J., Ward, L. M., Matzke, N. J. & Fischer, W. W. Crown group Oxyphotobacteria postdate the rise of oxygen. Geobiology 15, 19-29 (2017). https://doi.org:10.1111/gbi.12200
2. Gray, M. W., Burger, G. & Lang, B. F. Mitochondrial evolution. Science 283, 1476-1481 (1999).
3. Yoon, H. S., Hackett, J. D., Ciniglia, C., Pinto, G. & Bhattacharya, D. A molecular timeline for the origin of photosynthetic eukaryotes. Molecular biology and evolution 21, 809-818 (2004).
4. Margulis, L. & Sagan, D. What is life? , (1995).
5. Archibald, J. M. The puzzle of plastid evolution. Curr Biol 19, R81-88 (2009). https://doi.org:10.1016/j.cub.2008.11.067
6. Dorrell, R. G. & Howe, C. J. What makes a chloroplast? Reconstructing the establishment of photosynthetic symbioses. J Cell Sci 125, 1865-1875 (2012). https://doi.org:10.1242/jcs.102285
7. Melo Clavijo, J., Gould, S. B. & Christa, G. in Endosymbiotic Organelle Acquisition: Solutions to the Problem of Protein Localization and Membrane Passage 239-258 (Springer, 2024).
8. Maeda, T. et al. Chloroplast acquisition without the gene transfer in kleptoplastic sea slugs, Plakobranchus ocellatus. Elife 10 (2021). https://doi.org:10.7554/eLife.60176
9. Lhee, D., Bhattacharya, D. & Yoon, H. S. in Endosymbiotic Organelle Acquisition: Solutions to the Problem of Protein Localization and Membrane Passage (eds Steven D. Schwartzbach, Peter G. Kroth, & Miroslav Oborník) 3-38 (Springer International Publishing, 2024).
10. Altenburger, A. et al. Limits to the cellular control of sequestered cryptophyte prey in the marine ciliate Mesodinium rubrum. Isme j 15, 1056-1072 (2021). https://doi.org:10.1038/s41396-020-00830-9
11. Onuma, R. et al. Changes in the transcriptome, ploidy, and optimal light intensity of a cryptomonad upon integration into a kleptoplastic dinoflagellate. Isme j 14, 2407-2423 (2020). https://doi.org:10.1038/s41396-020-0693-4
12. Hehenberger, E., Gast, R. J. & Keeling, P. J. A kleptoplastidic dinoflagellate and the tipping point between transient and fully integrated plastid endosymbiosis. Proceedings of the National Academy of Sciences of the United States of America 116, 17934-17942 (2019). https://doi.org:10.1073/pnas.1910121116
13. Karnkowska, A. et al. Euglenozoan kleptoplasty illuminates the early evolution of photoendosymbiosis. Proceedings of the National Academy of Sciences of the United States of America 120, e2220100120 (2023). https://doi.org:10.1073/pnas.2220100120
14. Archibald, J. M., Rogers, M. B., Toop, M., Ishida, K. & Keeling, P. J. Lateral gene transfer and the evolution of plastid-targeted proteins in the secondary plastid-containing alga Bigelowiella natans. Proceedings of the National Academy of Sciences of the United States of America 100, 7678-7683 (2003). https://doi.org:10.1073/pnas.1230951100
15. Frommolt, R. et al. Ancient recruitment by chromists of green algal genes encoding enzymes for carotenoid biosynthesis. Mol Biol Evol 25, 2653-2667 (2008). https://doi.org:10.1093/molbev/msn206
16. Hackett, J. D. et al. Migration of the plastid genome to the nucleus in a peridinin dinoflagellate. Curr Biol 14, 213-218 (2004). https://doi.org:10.1016/j.cub.2004.01.032
17. Nosenko, T. & Bhattacharya, D. Horizontal gene transfer in chromalveolates. BMC evolutionary biology 7, 173 (2007). https://doi.org:10.1186/1471-2148-7-173
18. Nosenko, T. et al. Chimeric plastid proteome in the Florida "red tide" dinoflagellate Karenia brevis. Mol Biol Evol 23, 2026-2038 (2006). https://doi.org:10.1093/molbev/msl074
19. Larkum, A. W., Lockhart, P. J. & Howe, C. J. Shopping for plastids. Trends Plant Sci 12, 189-195 (2007). https://doi.org:10.1016/j.tplants.2007.03.011
20. Howe, C. J., Barbrook, A. C., Nisbet, R. E., Lockhart, P. J. & Larkum, A. W. The origin of plastids. Philosophical transactions of the Royal Society of London. Series B, Biological sciences 363, 2675-2685 (2008). https://doi.org:10.1098/rstb.2008.0050
21. Morozov, A. A. & Galachyants, Y. P. Diatom genes originating from red and green algae: Implications for the secondary endosymbiosis models. Marine genomics 45, 72-78 (2019). https://doi.org:10.1016/j.margen.2019.02.003
22.Chen, P. et al. A plant-derived natural photosynthetic system for improving cell anabolism. Nature 612, 546-554 (2022). https://doi.org:10.1038/s41586-022-05499-y










