不只双螺旋,DNA结构的多样性和演化
01
DNA结构的多样性
在格里菲斯(Frederick Griffith)和艾弗里(Osward Avery)等人的肺炎链球菌转化实验确定了DNA是生物体遗传信息的载体后,沃森(James D. Watson)和克里克(Francis Crick)等人于1953年揭示了DNA的二级结构模型——右手双螺旋结构(Double helix structure,又称B型DNA)(图一A),两条反向平行的DNA链螺旋盘绕,磷酸基团构成DNA链的骨架,内侧的碱基根据互补配对原则通过氢键结合1。自此,DNA的双螺旋结构深入人心,逐渐成为代表生命科学的符号。
实际上,DNA除了双螺旋结构,还可以形成其他的非B型DNA结构(图一B-G),包括左手双螺旋的Z DNA(B)、鸟嘌呤富集区域形成的G-四链体(G-quadruplex)(C)、DNA串联重复错配形成的Slipped DNA(D)、高GC含量的镜像重复区形成的三链DNA结构(H DNA)(E)、倒转重复序列两条臂(Arm)互补配对形成的发夹结构(F)和十字架结构(G)2。此外,胞嘧啶富集的序列在酸性条件下可能形成四链的i-motif结构。据估计,这些非B型DNA结构占人类基因组总长度的13%左右,结构的多样性使其能在细胞中发挥多样的功能,因而也受到不同的自然选择作用3。
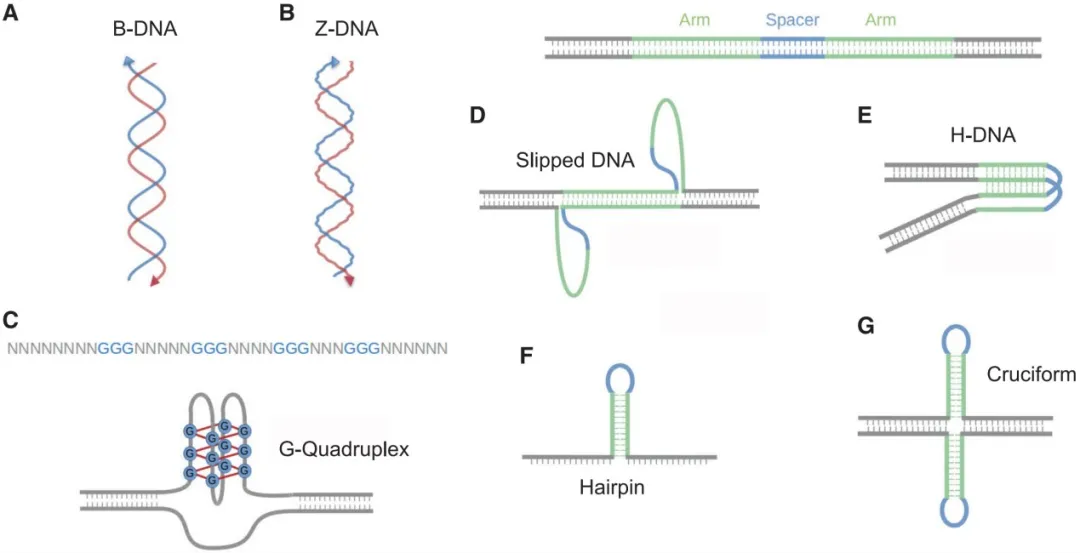
图一、双螺旋B型DNA和其他非B型DNA。图片来自引文2
02
什么是G-四链体?
G-四链体(简称G4)是研究最为广泛的非B型DNA,由四个鸟嘌呤(Guanine)通过Hoogsteen氢键连接形成的G-四分体(G-quartet)堆叠而成。对G4的研究最早可以追溯到1910年,德国化学家Ivar Bang发现高浓度的富含鸟嘌呤核苷酸的序列能形成凝胶,这一不同寻常的性质困惑了科学界长达50年之久4。直到Gellert等人通过X射线衍射技术发现鸟嘌呤能通过氢键连接形成G-四分体平面结构并进一步堆叠形成三维的G4结构,从而具有凝胶状的特性,这一谜题才被解开5。
03
G-四链体的分布和功能两面性
G4在从微生物到人类的细胞中广泛存在,其总长度约占人类基因组的1%,分布在端粒、启动子、增强子等区域6。G4在基因组中有着双刃剑的作用(图二):一方面,端粒区的G4结构能抑制核酸酶的活性,防止端粒被核酶降解7。启动子区形成的G4是转录因子结合的热点区域,进而增强下游基因的转录。例如,原癌基因MYC启动子区的G4对于招募转录因子和染色质调节蛋白是至关重要的,CRISPR技术破坏G4结构的形成会显著降低MYC的表达水平。近日,中国科学院长春应用化学研究所曲晓刚研究员团队将CRISPR与G4结合蛋白/配体结合,开发出特异性靶向G4的方法进而改变目标基因的表达水平,表明G4是基因表达操纵的潜在靶标8。此外,DNA复制起点上游的G4能激活DNA复制复合物,起始DNA的复制过程。另一方面,在DNA复制的过程中,富含鸟嘌呤的DNA区域可能形成G4,形成空间位阻阻碍DNA聚合酶的通过,进而抑制DNA的复制并增加复制错配和双链断裂的概率,最终诱导突变的发生。有研究表明G4的形成是癌细胞中基因组结构变异产生的关键因素9。
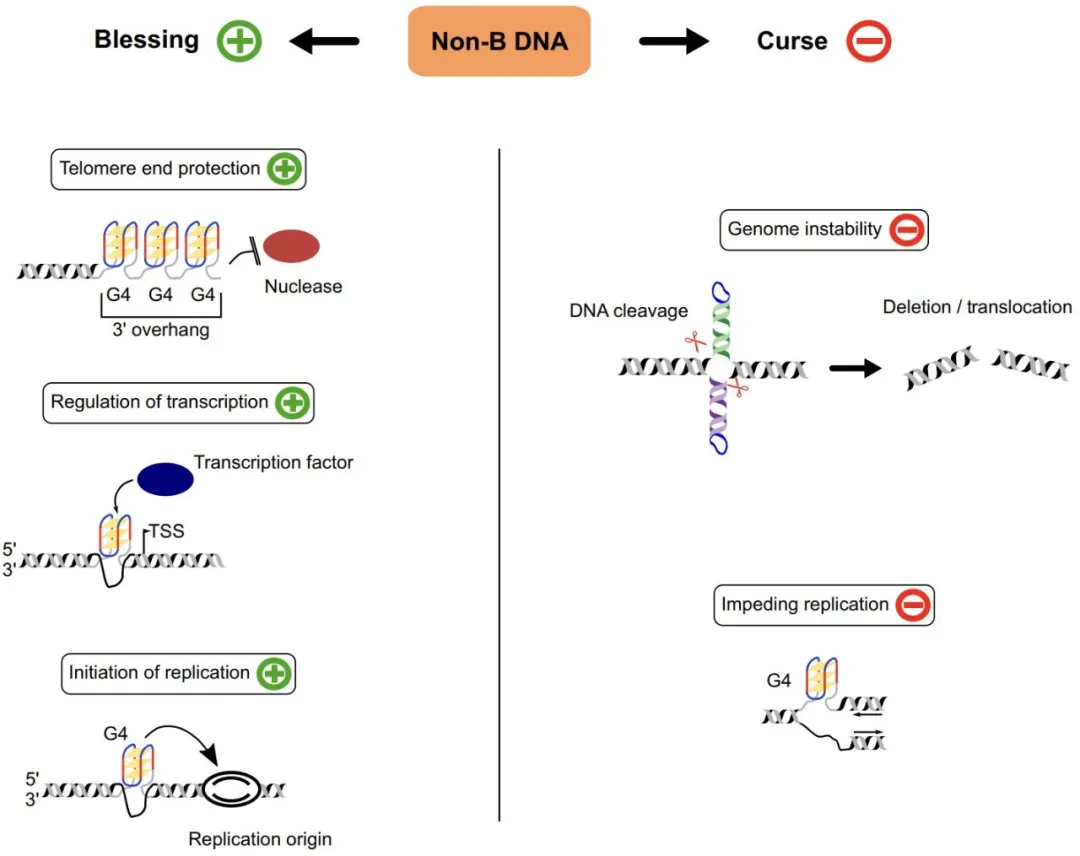
图二、G4在基因组中的双重作用。左侧代表G4的有益作用(Blessing),如保护端粒、招募转录因子调控基因表达和促进复制起始;右侧表明G4的有害作用(Curse),如复制过程中G4结构形成物理空间位阻可能阻碍DNA聚合酶的通过。图片来自引文7。
04
G-四链体的演化及选择压力
有功能的基因元件往往会受到负向选择在基因组中保留,而有害的元件通常会被自然选择清除。G4在基因组中的双重角色使其可能受到两种截然相反的选择作用。在病毒中,基于G4 motif分析结果显示感染原核和真核生物的病毒基因组对G4的选择偏好不同,主要感染原核生物的病毒基因组倾向于保留序列较短和稳定性较低的G4,而主要感染真核生物的病毒基因组则偏好结构更加稳定的G410。这可能是由于病毒基因组复制过程中G4的解链需要借助宿主的解旋酶和能量,而原核宿主的细胞中酶系统比较简单,只能解链结构稳定性较低的G4,因而感染原核细胞的病毒倾向保留结构稳定性较低的G4。
一项横跨多个类群物种的研究发现简单生命系统的核基因组中G4密度较低,而在生命系统更为复杂的物种中,G4的密度更高、单个G4的长度也更长11。线粒体中G4的演化趋势与核基因组相同(图三),G4的数量和密度在演化过程中均呈现出逐渐增加的趋势(图三、四)12。例如,鸟类和灵长类的G4密度显著高于昆虫和鱼类(图四)。考虑到GC含量可能影响G4的密度(图五),用GC含量校正后的G4密度也观察到相似的结果11,表明G4密度的增加不是GC含量的升高导致的,可能是自然选择驱动的结果。同时,基因组中不同区域对G4的选择也存在偏好6,7。启动子、CpG岛和DNA复制起始位点等功能区的G4密度较高,而重组热点区域和染色质拓扑结构域(TAD)边界的G4受到纯化选择作用较强,外显子区对应的非转录链上的G4受到纯化选择作用较弱、密度和热稳定性也较低,表明G4不倾向于在RNA分子上产生。因此,不同物种中的酶系统的复杂程度、基因组不同区域的功能及位置共同决定了G4受到的演化压力。
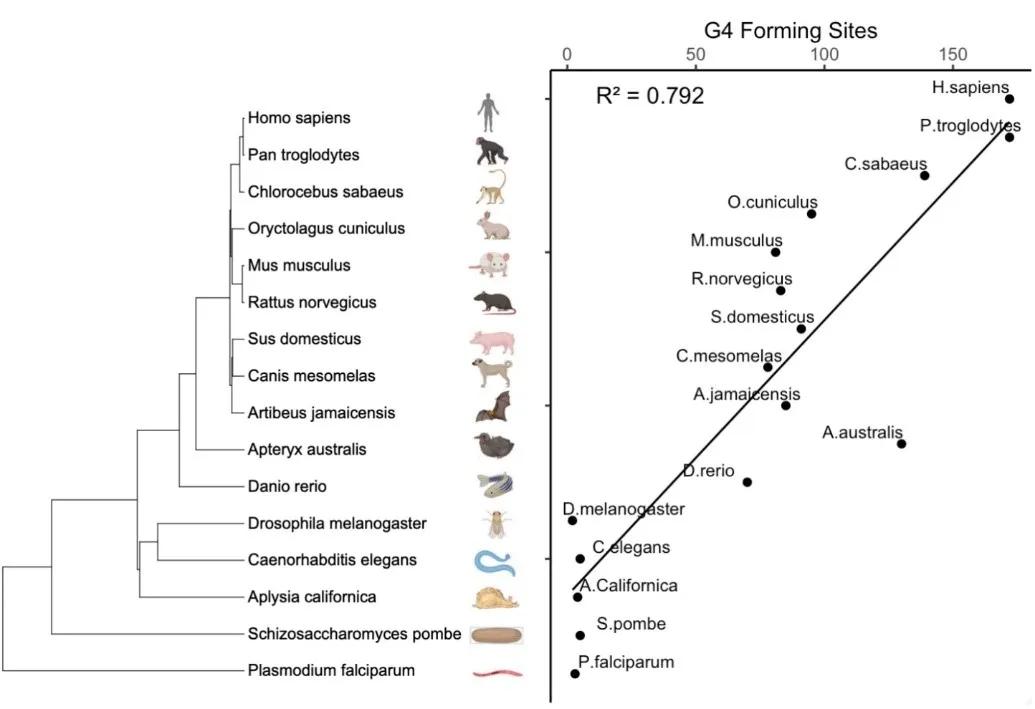
图三、不同物种线粒体DNA中G4的数量。图中数据来自引文12。
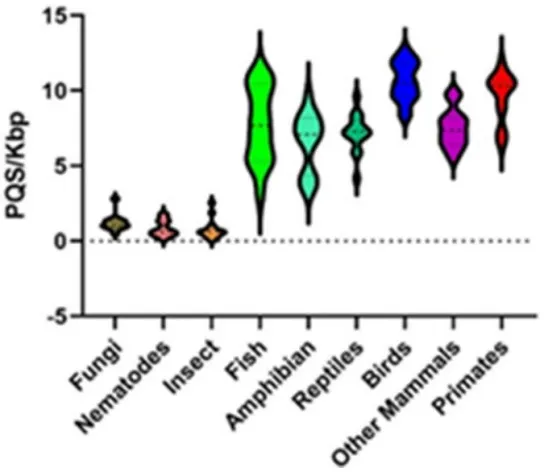
图四、不同类群的物种线粒体DNA的G4密度。PQS代表预测的G4。图片来自引文12。
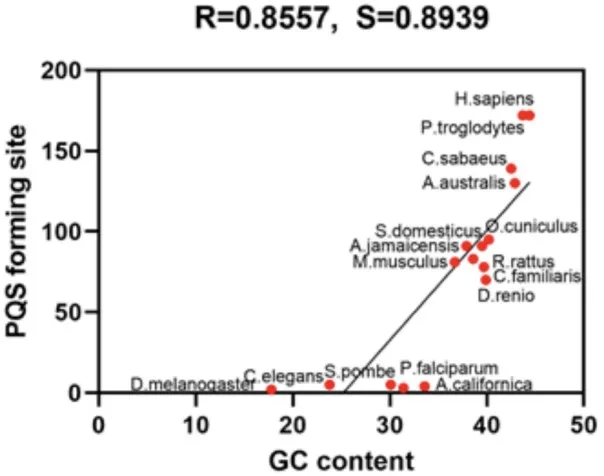
图五、不同物种线粒体DNA的GC含量和G4丰度的相关性。R和S分别表示Pearson和Spearman相关性。图片来自引文12。
05
总结
遗传物质的多样性是物种适应环境变化的重要保证。同样地,生物大分子的结构往往决定其生理特性和功能,DNA结构的多样性也是遗传物质多样性的重要组成成分,有助于物种对环境的适应。然而,由于缺乏有效的鉴定方法,非B型DNA在基因组中的功能长期被忽略。G4作为其中的代表,大部分关于G4的研究也主要依赖于基于序列的预测方法,其结果与细胞中真实存在的G4可能存在较大差异,关于G4的功能和演化模式还需要未来更多更直接的证据。
双螺旋结构万口传,至今已觉不新鲜;
DNA新构象代代出,各领风骚数百年。
参考文献
1.Watson J D, Crick F H C. Molecular structure of nucleic acids: a structure for deoxyribose nucleic acid[J]. Nature, 1953, 171(4356): 737-738.
2. Georgakopoulos-Soares I, Victorino J, Parada G E, et al. High-throughput characterization of the role of non-B DNA motifs on promoter function[J]. Cell genomics, 2022, 2(4).
3. Guiblet W M, Cremona M A, Cechova M, et al. Long-read sequencing technology indicates genome-wide effects of non-B DNA on polymerization speed and error rate[J]. Genome Research, 2018, 28(12): 1767-1778.
4. Bang I. Untersuchungen über die Guanylsäure[J]. Biochem. Z, 1910, 26: 293-311.
5. Gellert M, Lipsett M N, Davies D R. Helix formation by guanylic acid[J]. Proceedings of the National Academy of Sciences, 1962, 48(12): 2013-2018.
6. Guiblet W M, DeGiorgio M, Cheng X, et al. Selection and thermostability suggest G-quadruplexes are novel functional elements of the human genome[J]. Genome research, 2021, 31(7): 1136-1149.
7. Makova K D, Weissensteiner M H. Noncanonical DNA structures are drivers of genome evolution[J]. Trends in Genetics, 2023, 39(2): 109-124.
8. Qin G, Liu Z, Yang J, et al. Targeting specific DNA G-quadruplexes with CRISPR-guided G-quadruplex-binding proteins and ligands[J]. Nature Cell Biology, 2024: 1-13.
9. Zhang R, Shu H, Wang Y, et al. G-quadruplex structures are key modulators of somatic structural variants in cancers[J]. Cancer Research, 2023, 83(8): 1234-1248.
10. Li Z, Qian SH, Wang F, et al. G-quadruplexes in genomes of viruses infecting eukaryotes or prokaryotes are under different selection pressures from hosts[J]. Journal of Genetics and Genomics, 2022, 49(1): 20-29.
11. Wu F, Niu K, Cui Y, et al. Genome-wide analysis of DNA G-quadruplex motifs across 37 species provides insights into G4 evolution[J]. Communications biology, 2021, 4(1): 98.
12. Sahayasheela V J, Yu Z, Hidaka T, et al. Mitochondria and G-quadruplex evolution: an intertwined relationship[J]. Trends in Genetics, 2023, 39(1): 15-30.










